Sperling Nội tiết học Nhi khoa, Ấn bản thứ 5 – Biên dịch: Ths.Bs. Lê Đình Sáng
Sperling Pediatric Endocrinology, Fifth Edition
Tác giả: Sperling, Mark A., MD – Nhà xuất bản: Elsevier Inc.
PHẦN I: CÁC NGUYÊN LÝ VÀ PHƯƠNG PHÁP CỦA NỘI TIẾT HỌC NHI KHOA
CHƯƠNG 2: NỘI TIẾT HỌC PHÂN TỬ, DI TRUYỀN HỌC NỘI TIẾT, VÀ Y HỌC CHÍNH XÁC
Molecular Endocrinology, Endocrine Genetics, and Precision Medicine
Ram K. Menon; Massimo Trucco; Joseph A. Majzoub; Constantine A. Stratakis
Sperling Pediatric Endocrinology, 2, 9-29
GIỚI THIỆU
Nghiên cứu về hệ nội tiết đã trải qua một sự phát triển vượt bậc kể từ những năm 1990, từ các nghiên cứu sinh lý học truyền thống chiếm ưu thế trong nhiều năm đến những khám phá của nội tiết học phân tử (molecular endocrinology) và di truyền học nội tiết (endocrine genetics). Hiện tại, tác động lớn của y học phân tử đối với thực hành nội tiết nhi khoa liên quan đến chẩn đoán và tư vấn di truyền cho nhiều rối loạn nội tiết di truyền khác nhau. Ngược lại, việc áp dụng trực tiếp kiến thức mới này vào điều trị vẫn còn ở giai đoạn sơ khai. Ung thư học nội tiết đã được hưởng lợi rất nhiều từ việc áp dụng các loại thuốc mới được thiết kế để nhắm vào các đột biến cụ thể, ví dụ như trong ung thư tuyến giáp. Một tiến bộ điều trị đáng chú ý gần đây sau khi xác định được cơ sở phân tử của một rối loạn nội tiết là sự phát triển của kháng thể đơn dòng burosumab nhắm vào protein yếu tố tăng trưởng nguyên bào sợi-23 để điều trị bệnh còi xương hạ phosphat máu liên kết nhiễm sắc thể X (X-linked hypophosphatemic rickets). Chương này giới thiệu các nguyên tắc cơ bản của sinh học phân tử, các kỹ thuật phòng thí nghiệm phổ biến, và một số ví dụ về những tiến bộ gần đây trong các rối loạn nội tiết nhi khoa lâm sàng với trọng tâm là di truyền học nội tiết. Hầu hết các xét nghiệm chẩn đoán, dược lý di truyền, và liệu pháp phân tử mới đều được thảo luận trong các chương cụ thể về bệnh của cuốn sách này, và chỉ những ví dụ làm nổi bật nguyên tắc/chiến lược đang được thảo luận mới được đề cập trong chương này.
CÁC CÔNG CỤ PHÂN TỬ CƠ BẢN
Phân lập và Cắt DNA và Thấm Southern (Southern Blotting)
Nhiễm sắc thể của con người bao gồm một phân tử xoắn kép dài của acid deoxyribonucleic (DNA) (deoxyribonucleic acid) liên kết với các protein nhân khác nhau. Vì DNA là điểm khởi đầu của quá trình tổng hợp tất cả các phân tử protein trong cơ thể, các kỹ thuật phân tử sử dụng DNA đã chứng tỏ vai trò quan trọng trong việc phát triển các công cụ chẩn đoán để phân tích các bệnh nội tiết. DNA có thể được phân lập từ bất kỳ mô nào của con người, bao gồm cả các tế bào bạch cầu lưu thông. Khoảng 200 µg DNA có thể được thu nhận từ 10 đến 20 mL máu toàn phần, với hiệu quả chiết xuất DNA phụ thuộc vào kỹ thuật được sử dụng và phương pháp chống đông. DNA đã chiết xuất có thể được lưu trữ gần như vô thời hạn ở nhiệt độ thích hợp. Hơn nữa, các tế bào lympho có thể được biến đổi bằng virus Epstein-Barr (hoặc các phương tiện khác) để tăng sinh vô hạn trong nuôi cấy tế bào thành các dòng tế bào “bất tử”, do đó cung cấp một nguồn DNA tái tạo. Để thực hiện các nghiên cứu di truyền phân tử, các dòng tế bào lympho biến đổi thường là mô được lựa chọn, bởi vì một nguồn DNA tái tạo giúp loại bỏ nhu cầu lấy thêm máu từ gia đình. Các dòng nuôi cấy có nguồn gốc từ nguyên bào sợi cũng có thể đóng vai trò là nguồn DNA hoặc acid ribonucleic (RNA) (ribonucleic acid) vĩnh viễn (một khi đã được biến đổi), nhưng chúng phải được lấy từ các mẫu phẫu thuật hoặc sinh thiết. Cần lưu ý rằng, vì sự biểu hiện của nhiều gen là đặc hiệu cho từng mô, các dòng tế bào lympho hoặc nguyên bào sợi bất tử không thể được sử dụng để phân tích sự phong phú hoặc thành phần của RNA thông tin (mRNA) (messenger RNA) cho một gen cụ thể. Do đó, các nghiên cứu liên quan đến mRNA đòi hỏi phải phân tích (các) mô biểu hiện gen đó, như được nêu trong phần “Phân tích RNA”. Gần đây, vấn đề về lượng DNA hạn chế có thể thu được từ một số nguồn nhất định đã được khắc phục bằng việc sử dụng phản ứng chuỗi polymerase (PCR) (polymerase chain reaction), một phương pháp linh hoạt để nhân lên một cách trung thực các đoạn DNA ban đầu.
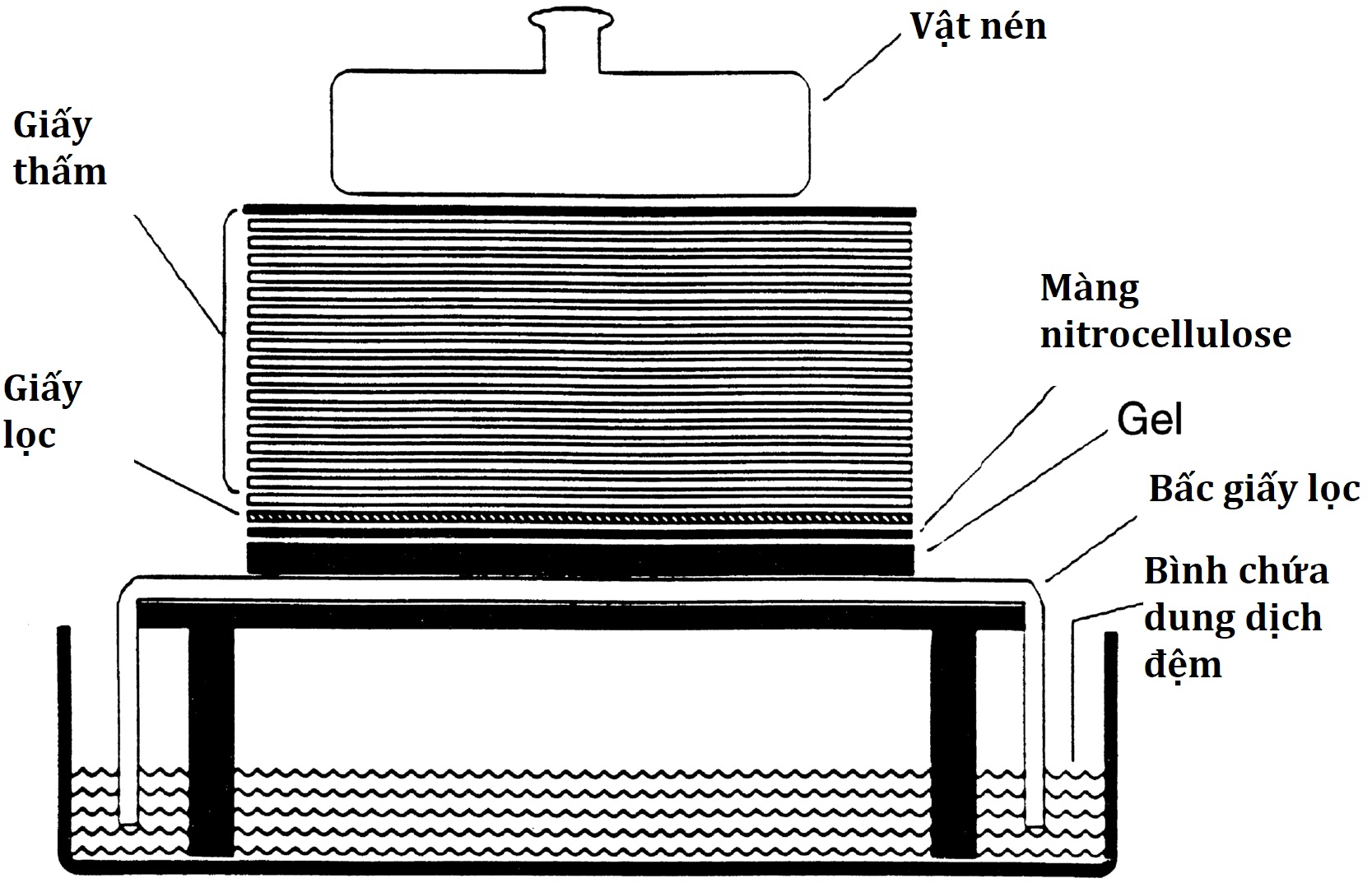 Hình 2.1 Thấm Southern. Các đoạn DNA sợi đôi được phân tách theo kích thước bằng điện di trên gel agarose. Để làm cho DNA trở thành sợi đơn (biến tính), gel agarose được ngâm trong dung dịch acid. Sau khi trung hòa acid, gel được đặt lên giấy lọc, các đầu của giấy lọc nằm trong một bể chứa dung dịch đệm muối đậm đặc. Một tấm màng nitrocellulose được đặt lên trên gel và giấy thấm được xếp chồng lên trên màng nitrocellulose. Dung dịch muối được hút lên qua gel nhờ tác động mao dẫn của bấc giấy lọc và giấy thấm. Khi dung dịch muối di chuyển qua gel, nó mang theo các đoạn DNA. Vì nitrocellulose liên kết với DNA sợi đơn, các đoạn DNA được lắng đọng lên nitrocellulose theo đúng khuôn mẫu mà chúng đã được đặt trong gel agarose. Các đoạn DNA liên kết với nitrocellulose được cố định vào màng bằng nhiệt hoặc tia cực tím. Màng nitrocellulose với DNA đã liên kết sau đó có thể được sử dụng cho các quy trình, chẳng hạn như lai với một mẫu dò DNA được đánh dấu. Các kỹ thuật chuyển DNA sang các chất nền liên kết khác, chẳng hạn như nylon, cũng tương tự.
Hình 2.1 Thấm Southern. Các đoạn DNA sợi đôi được phân tách theo kích thước bằng điện di trên gel agarose. Để làm cho DNA trở thành sợi đơn (biến tính), gel agarose được ngâm trong dung dịch acid. Sau khi trung hòa acid, gel được đặt lên giấy lọc, các đầu của giấy lọc nằm trong một bể chứa dung dịch đệm muối đậm đặc. Một tấm màng nitrocellulose được đặt lên trên gel và giấy thấm được xếp chồng lên trên màng nitrocellulose. Dung dịch muối được hút lên qua gel nhờ tác động mao dẫn của bấc giấy lọc và giấy thấm. Khi dung dịch muối di chuyển qua gel, nó mang theo các đoạn DNA. Vì nitrocellulose liên kết với DNA sợi đơn, các đoạn DNA được lắng đọng lên nitrocellulose theo đúng khuôn mẫu mà chúng đã được đặt trong gel agarose. Các đoạn DNA liên kết với nitrocellulose được cố định vào màng bằng nhiệt hoặc tia cực tím. Màng nitrocellulose với DNA đã liên kết sau đó có thể được sử dụng cho các quy trình, chẳng hạn như lai với một mẫu dò DNA được đánh dấu. Các kỹ thuật chuyển DNA sang các chất nền liên kết khác, chẳng hạn như nylon, cũng tương tự.
DNA tồn tại dưới dạng các phân tử cực lớn; nhiễm sắc thể thường nhỏ nhất (nhiễm sắc thể 22) có khoảng 50 triệu cặp base và toàn bộ bộ gen đơn bội của con người được ước tính bao gồm 3 đến 4 tỷ cặp base. Kích thước cực lớn này ngăn cản việc phân tích DNA ở dạng tự nhiên trong các kỹ thuật sinh học phân tử thông thường. Các kỹ thuật nhận dạng và phân tích DNA đã trở nên khả thi và dễ tiếp cận với việc phát hiện ra các enzyme được gọi là endonuclease giới hạn (restriction endonucleases). Các enzyme này, ban đầu được phân lập từ vi khuẩn, cắt DNA thành các kích thước nhỏ hơn dựa trên các vị trí nhận biết cụ thể dài từ hai đến tám cặp base. Thuật ngữ giới hạn đề cập đến chức năng của các enzyme này trong vi khuẩn. Một endonuclease giới hạn phá hủy DNA ngoại lai (chẳng hạn như DNA của thể thực khuẩn) bằng cách cắt DNA tại các vị trí cụ thể, do đó “hạn chế” sự xâm nhập của DNA ngoại lai vào vi khuẩn. Hàng trăm enzyme giới hạn với các vị trí nhận biết khác nhau hiện có sẵn trên thị trường. Vì vị trí nhận biết của một enzyme nhất định là cố định, số lượng và kích thước của các đoạn được tạo ra cho một phân tử DNA cụ thể vẫn nhất quán với số lượng vị trí nhận biết và cung cấp các khuôn mẫu có thể dự đoán được sau khi phân tách bằng điện di.
Việc phân tích vài trăm cặp base DNA trong vùng quan tâm là rất khó khăn khi DNA từ tất cả các nhiễm sắc thể của con người được cắt và phân tách trên cùng một gel. Những hạn chế này được khắc phục bằng kỹ thuật thấm Southern (Southern blotting) (được đặt theo tên của người phát minh ra nó, Edward Southern). Thấm Southern bao gồm việc cắt DNA và phân tách bằng điện di trên agarose. Sau khi điện di, DNA được chuyển sang một giá đỡ rắn (chẳng hạn như màng nitrocellulose hoặc nylon), cho phép sao chép khuôn mẫu của các đoạn DNA đã phân tách lên màng (Hình 2.1). Sau đó, DNA được biến tính (tức là hai sợi được tách rời vật lý), cố định vào màng, và màng khô được trộn với dung dịch chứa mẫu dò DNA. Mẫu dò DNA (DNA probe) là một đoạn DNA chứa một chuỗi nucleotide đặc hiệu cho gen hoặc vùng nhiễm sắc thể quan tâm. Để phát hiện, mẫu dò DNA được đánh dấu bằng một thẻ nhận dạng, chẳng hạn như phốt pho phóng xạ (ví dụ ) hoặc một gốc phát quang hóa học; loại sau hiện nay đã gần như thay thế hoàn toàn phóng xạ. Quá trình trộn mẫu dò DNA với DNA biến tính được cố định trên màng được gọi là lai hóa (hybridization), nguyên tắc là chỉ có bốn base acid nucleic trong DNA—adenine (A), thymidine (T), guanine (G), và cytosine (C)—luôn bổ sung cho nhau trên hai sợi DNA, A bắt cặp với T, và G bắt cặp với C. Sau khi lai hóa, màng được rửa để loại bỏ mẫu dò không liên kết và được phơi sáng với phim X-quang trong một quá trình gọi là chụp tự ghi phóng xạ (radioautography, còn gọi là autoradiography) để phát hiện phốt pho phóng xạ hoặc trong một quá trình được sử dụng để phát hiện thẻ phát quang hóa học. Chỉ những đoạn bổ sung và đã liên kết với mẫu dò chứa DNA quan tâm mới hiện rõ trên phim X-quang, cho phép phân tích kích thước và khuôn mẫu của các đoạn này. Theo quy trình thông thường, kỹ thuật phân tích Southern có thể phát hiện một gen đơn bản trong ít nhất 5 µg DNA, tương đương với hàm lượng DNA của khoảng 10 tế bào.
Đa hình Chiều dài Đoạn cắt Giới hạn (Restriction Fragment Length Polymorphism – RFLP)
Đa hình chiều dài đoạn cắt giới hạn (RFLP) (Restriction fragment length polymorphism) là một kỹ thuật hiện nay hiếm khi được sử dụng nhưng lại xuất hiện rộng rãi trong y văn di truyền học nội tiết, vì một số khám phá di truyền nội tiết trong 2 đến 3 thập kỷ qua đã dựa trên kỹ thuật này. Số lượng và kích thước của các đoạn DNA do việc cắt một vùng DNA cụ thể tạo thành một khuôn mẫu có thể nhận biết được. Những biến thể nhỏ trong trình tự giữa các cá thể không có quan hệ họ hàng có thể làm cho một vị trí nhận biết của enzyme giới hạn có mặt hoặc vắng mặt; điều này dẫn đến sự thay đổi về số lượng và khuôn mẫu kích thước của các đoạn DNA được tạo ra bởi việc cắt bằng enzyme đó. Do đó, vùng này được cho là đa hình đối với enzyme cụ thể được thử nghiệm, tức là một RFLP (Hình 2.2). Giá trị của RFLP là nó có thể được sử dụng như một thẻ phân tử để theo dõi sự di truyền của các alen từ mẹ và cha. Hơn nữa, vùng đa hình được phân tích không cần phải mã hóa cho biến thể di truyền là nguyên nhân của bệnh đang được nghiên cứu, mà chỉ cần nằm gần gen quan tâm. Khi một khuôn mẫu RFLP cụ thể có thể được chứng minh là có liên quan đến một bệnh, việc so sánh khuôn mẫu RFLP của con cái với khuôn mẫu RFLP của cha mẹ bị ảnh hưởng hoặc mang gen có thể xác định khả năng một người con thừa hưởng bệnh. Hạn chế chính của kỹ thuật RFLP là khả năng ứng dụng của nó để phân tích bất kỳ gen cụ thể nào phụ thuộc vào kiến thức trước đó về sự hiện diện của các vị trí giới hạn đa hình thuận tiện (“có thông tin”) nằm cạnh gen quan tâm trong khoảng tối đa vài kilobase. Vì những tiêu chí này có thể không được đáp ứng trong bất kỳ trường hợp nào, khả năng ứng dụng của RFLP không thể được đảm bảo cho việc phân tích một gen nhất định.
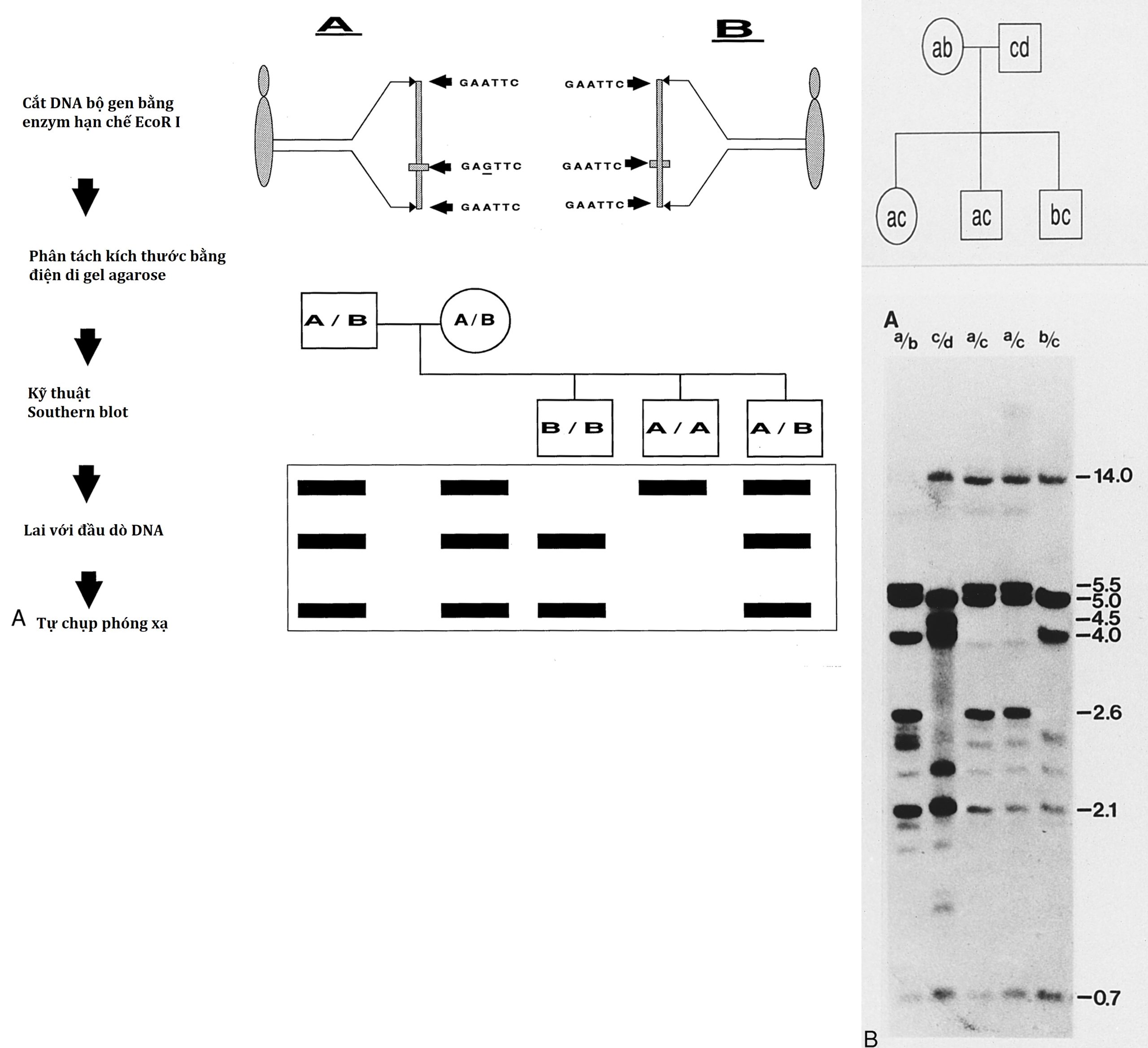
Hình 2.2 Đa hình Chiều dài Đoạn cắt Giới hạn (RFLP). A, Minh họa sơ đồ. A và B đại diện cho hai alen hiển thị một vị trí đa hình cho enzyme giới hạn EcoR I. EcoR I sẽ cắt DNA có trình tự “GAATTC”; do đó, alen B sẽ bị EcoR I cắt tại ba vị trí để tạo ra hai đoạn DNA, trong khi alen A sẽ chỉ bị EcoR I cắt hai lần và không cắt tại vị trí (được chỉ định bằng thanh ngang) nơi nucleotide G (gạch chân) thay thế nucleotide A có trong alen B. Sau khi cắt, DNA được phân đoạn theo kích thước bằng điện di trên gel agarose và chuyển sang màng bằng kỹ thuật thấm Southern (xem Hình 2.1 để biết chi tiết). Sau đó, màng được lai với một mẫu dò DNA được đánh dấu, chứa toàn bộ trình tự được bao phủ bởi ba vị trí EcoR I. Chụp tự ghi phóng xạ của màng sẽ phát hiện kích thước của các đoạn DNA được tạo ra bởi quá trình cắt bằng enzyme giới hạn. Trong minh họa cụ thể này, cả hai cha mẹ đều là dị hợp tử và sở hữu cả alen A và B. Việc khớp khuôn mẫu của các dải DNA của con cái với cha mẹ sẽ thiết lập mô hình di truyền của các alen. Ví dụ, nếu alen A đại diện cho alen bất thường của một bệnh lặn trên nhiễm sắc thể thường, thì việc kiểm tra thấm Southern sẽ xác định rằng (từ trái sang phải) người con đầu tiên (B/B) là đồng hợp tử cho alen bình thường, người con thứ hai (A/A) là đồng hợp tử cho alen bất thường, và người con thứ ba (A/B) là người mang gen. B, Phân tích RFLP của gen DQ-beta của locus kháng nguyên bạch cầu người (HLA). DNA bộ gen từ các thành viên của phả hệ được chỉ định đã được cắt bằng enzyme giới hạn Pst I, phân đoạn theo kích thước bằng điện di trên gel agarose, và chuyển sang màng nitrocellulose bằng kỹ thuật thấm Southern. Màng sau đó được lai với một mẫu dò DNA bổ sung đặc hiệu cho gen DQ-beta; mẫu dò dư thừa được loại bỏ bằng cách rửa ở độ nghiêm ngặt thích hợp và được phân tích bằng chụp tự ghi phóng xạ. Kích thước của các đoạn DNA (tính bằng kilobase, kb) được chỉ định ở bên phải. Biểu đồ phả hệ chỉ ra các alen đa hình (a, b, c, d) và các dải trên thấm Southern tương ứng với các alen này (a 5,5kb, b 5,0kb, c 14,0kb, d 4,5kb) cho thấy mô hình di truyền của các alen này.
Phản ứng Chuỗi Polymerase (Polymerase Chain Reaction – PCR)
PCR là một kỹ thuật được phát triển vào cuối những năm 1980 và đã cách mạng hóa sinh học phân tử (Hình 2.3). PCR cho phép khuếch đại logarit có chọn lọc một đoạn DNA mong muốn từ một hỗn hợp DNA phức tạp mà về mặt lý thuyết chứa ít nhất một bản sao của đoạn mục tiêu. Trong ứng dụng điển hình của kỹ thuật này, cần có một số kiến thức về trình tự DNA trong vùng cần khuếch đại, để có thể tổng hợp một cặp oligonucleotide ngắn (dài khoảng 18-25 base) đặc hiệu (“mồi” – primers). Các mồi được tổng hợp theo cách mà chúng xác định giới hạn của vùng cần khuếch đại. Khuôn DNA chứa đoạn cần khuếch đại được biến tính bằng nhiệt, sao cho các sợi được tách ra và sau đó được làm nguội để cho phép các mồi bắt cặp vào các vùng bổ sung tương ứng. Enzyme Taq polymerase, một enzyme bền nhiệt ban đầu được phân lập từ vi khuẩn Thermophilus aquaticus, sau đó được sử dụng để bắt đầu tổng hợp (kéo dài) DNA. DNA được biến tính, bắt cặp, và kéo dài lặp đi lặp lại trong các chu kỳ liên tiếp trong một máy gọi là máy luân nhiệt (thermocycler) cho phép quá trình này được tự động hóa. Trong xét nghiệm thông thường, các chu kỳ lặp đi lặp lại này của việc biến tính, bắt cặp, và kéo dài dẫn đến việc tổng hợp khoảng 1 triệu bản sao của vùng mục tiêu trong khoảng 2 giờ. Để xác định tính xác thực của quá trình khuếch đại, danh tính của DNA được khuếch đại có thể được phân tích bằng điện di, lai với mẫu dò RNA hoặc DNA, cắt bằng (các) enzyme giới hạn có thông tin, hoặc được giải trình tự DNA trực tiếp. Sự đơn giản tương đối kết hợp với sức mạnh của kỹ thuật này đã dẫn đến việc sử dụng rộng rãi quy trình này và đã tạo ra nhiều biến thể và sửa đổi khác nhau đã được phát triển cho các ứng dụng cụ thể. Từ quan điểm thực tế, nhược điểm chính của PCR là xu hướng bị nhiễm chéo DNA mục tiêu. Nhược điểm này là kết quả trực tiếp của độ nhạy cực cao của phương pháp cho phép khuếch đại từ một phân tử của khuôn DNA ban đầu. Do đó, việc chuyển giao không chủ ý các trình tự được khuếch đại sang các vật dụng được sử dụng trong quy trình sẽ khuếch đại DNA trong các mẫu không chứa trình tự DNA mục tiêu (tức là kết quả dương tính giả). Nhiễm chéo nên bị nghi ngờ khi sự khuếch đại xảy ra ở các mẫu đối chứng âm không chứa khuôn mục tiêu. Một trong những phương thức nhiễm chéo phổ biến nhất là thông qua việc tạo aerosol của DNA được khuếch đại trong các quy trình phòng thí nghiệm thông thường, chẳng hạn như vortex, pipet, và thao tác với các ống vi ly tâm. Sự cẩn thận tỉ mỉ trong kỹ thuật thực nghiệm, tổ chức đúng cách nơi làm việc PCR, và bao gồm các đối chứng thích hợp là điều cần thiết để ngăn chặn thành công sự nhiễm chéo trong các thí nghiệm PCR.
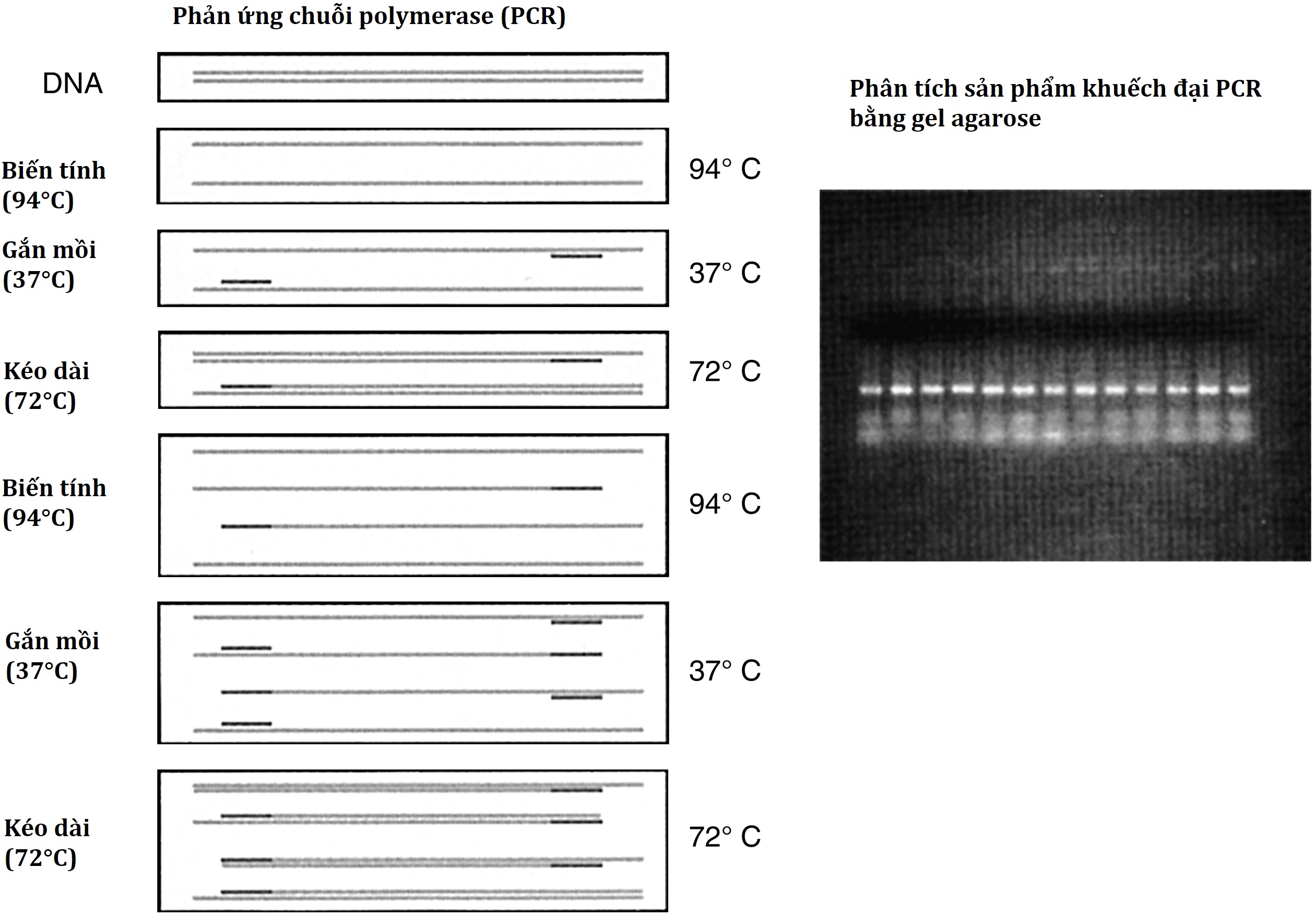
Hình 2.3 Phản ứng Chuỗi Polymerase (PCR). Một cặp mồi oligonucleotide (thanh đặc), bổ sung cho các trình tự nằm cạnh một vùng quan tâm cụ thể (thanh tô bóng, chấm), được sử dụng để dẫn hướng tổng hợp DNA theo các hướng đối diện và chồng chéo. Các chu kỳ lặp đi lặp lại của biến tính DNA, bắt cặp mồi, và tổng hợp DNA (kéo dài mồi) bởi enzyme DNA polymerase dẫn đến sự gia tăng theo cấp số nhân của DNA mục tiêu (tức là, trình tự DNA nằm giữa hai mồi) sao cho đoạn DNA này có thể được khuếch đại 1×10^6-7 lần sau 30 chu kỳ như vậy. Việc sử dụng DNA polymerase bền nhiệt (tức là, Taq polymerase) cho phép quy trình này được tự động hóa. Hình nhỏ: DNA được khuếch đại có thể được sử dụng để phân tích tiếp theo (tức là, phân đoạn theo kích thước bằng điện di trên gel agarose).
Nói chung, các ứng dụng PCR hoặc là hướng đến việc xác định một trình tự DNA cụ thể trong một mẫu mô hoặc dịch cơ thể hoặc được sử dụng để sản xuất một lượng DNA tương đối lớn của một trình tự cụ thể, sau đó được sử dụng trong các nghiên cứu sâu hơn. Các ví dụ về loại ứng dụng đầu tiên là phổ biến trong nhiều lĩnh vực y học, chẳng hạn như trong vi sinh học, trong đó kỹ thuật PCR được sử dụng để phát hiện sự hiện diện của các trình tự DNA đặc hiệu cho virus hoặc vi khuẩn trong một mẫu sinh học. Các ví dụ về một ứng dụng như vậy trong nội tiết nhi khoa bao gồm việc sử dụng PCR của gen SRY để phát hiện vật chất nhiễm sắc thể Y ở bệnh nhân có hội chứng Turner được xác định bằng kiểu nhân đồ và việc xác định nhanh chóng giới tính nhiễm sắc thể trong các trường hợp giới tính thai nhi hoặc sơ sinh không rõ ràng (Hình 2.4).
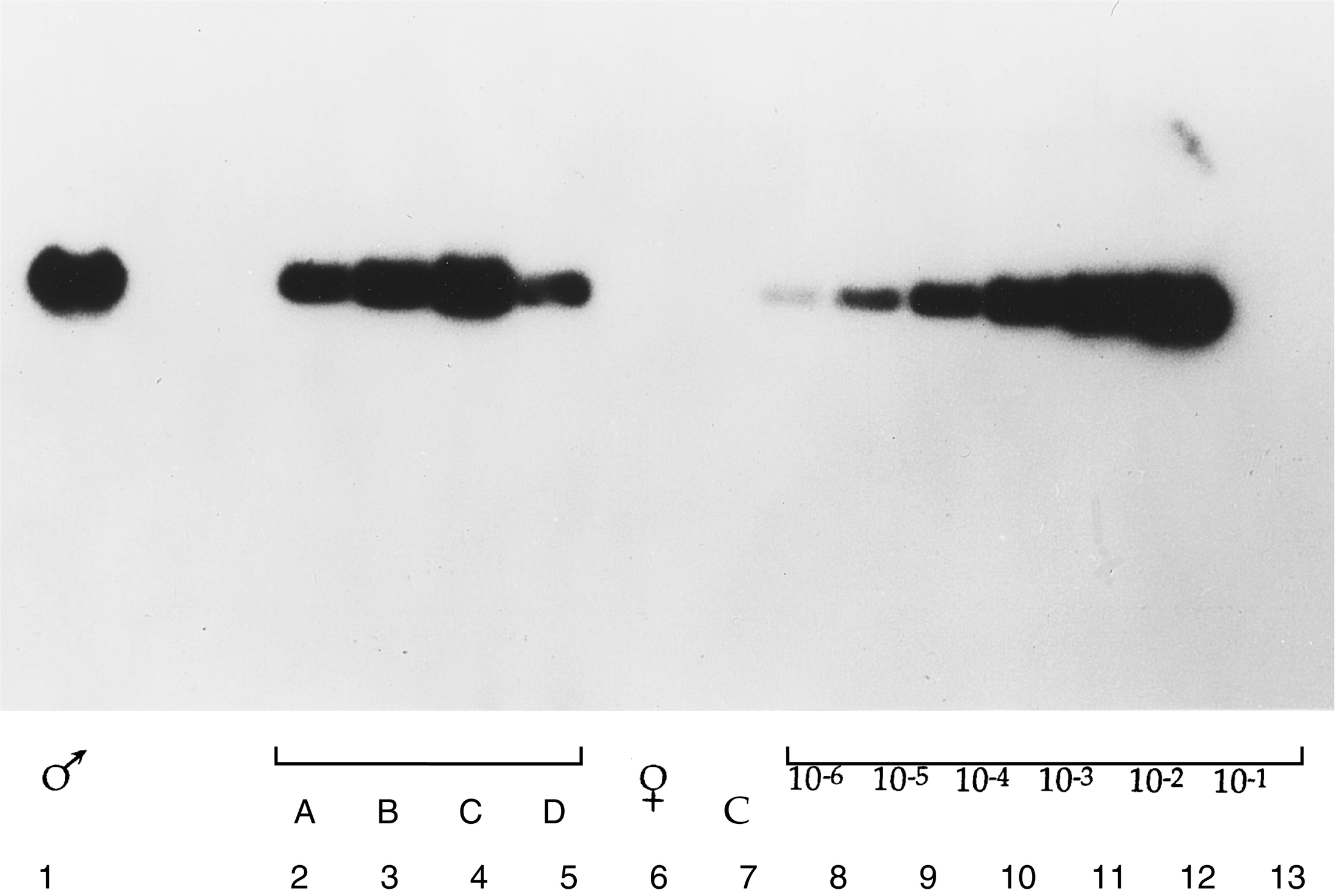
Hình 2.4 Phát hiện trình tự đặc hiệu gen SRY trong hội chứng Turner bằng khuếch đại phản ứng chuỗi polymerase (PCR) và thấm Southern. Các mồi đặc hiệu SRY đã được sử dụng trong PCR để khuếch đại DNA từ các bệnh nhân có kiểu nhân đồ 45,X. DNA được khuếch đại đã được phân đoạn theo kích thước bằng điện di trên gel-agarose và chuyển sang màng bằng kỹ thuật thấm Southern. Màng sau đó được lai với DNA đặc hiệu SRY được đánh dấu và chụp tự ghi phóng xạ. Từ trái sang phải: DNA nam được khuếch đại (giếng 1); DNA được khuếch đại từ các bệnh nhân có kiểu nhân đồ 45,X (giếng 2-5); DNA nữ được khuếch đại (giếng 6); đối chứng âm không có DNA (giếng 7); pha loãng nối tiếp DNA nam (giếng 8-13).
Hầu hết các ứng dụng PCR, cả dưới dạng công cụ nghiên cứu và sử dụng lâm sàng, đều hướng đến việc sản xuất DNA mục tiêu hoặc DNA bổ sung của một trình tự RNA mục tiêu. DNA được tạo ra (“khuếch đại”) sau đó được phân tích bằng các kỹ thuật khác, chẳng hạn như giải trình tự DNA.
Phân tích RNA
Phần lớn (>95%) DNA nhiễm sắc thể đại diện cho các trình tự không mã hóa. Các trình tự này chứa các yếu tố điều hòa, đóng vai trò là vị trí cho việc cắt nối thay thế, và chịu sự methyl hóa và các thay đổi ngoại di truyền khác ảnh hưởng đến chức năng gen. Tuy nhiên, hiện tại hầu hết các đột biến liên quan đến bệnh trong gen người đã được xác định trong các trình tự mã hóa. Một chiến lược thay thế để phân tích các đột biến trong một gen nhất định là nghiên cứu mRNA của nó, là sản phẩm (thông qua phiên mã) của 5% còn lại của DNA nhiễm sắc thể mã hóa cho protein. Ngoài ra, vì vốn mRNA là đặc hiệu cho từng tế bào và mô, việc phân tích các trình tự mRNA cung cấp thông tin độc đáo về các protein đặc hiệu cho mô được sản xuất trong một cơ quan/mô cụ thể.
Có nhiều kỹ thuật để phân tích mRNA. Kỹ thuật lâu đời nhất và được sử dụng rộng rãi nhất trong quá khứ, mặc dù hiện nay hiếm khi được sử dụng, là thấm Northern (Northern blotting) (được đặt tên như vậy vì nó dựa trên cùng nguyên tắc với thấm Southern), là một trong những phương pháp ban đầu được sử dụng để phân tích mRNA. Trong thấm Northern, RNA được biến tính bằng cách xử lý nó với một tác nhân, chẳng hạn như formaldehyde, để đảm bảo rằng RNA vẫn không gấp khúc và ở dạng tuyến tính. RNA biến tính sau đó được điện di và chuyển sang một giá đỡ rắn (chẳng hạn như màng nitrocellulose) theo cách tương tự như được mô tả cho thấm Southern. Màng với các phân tử RNA được phân tách theo kích thước được dò bằng mẫu dò DNA đặc hiệu cho gen được đánh dấu bằng một thẻ nhận dạng, như trong trường hợp thấm Southern, là một nhãn phóng xạ (ví dụ, 32P) hoặc phổ biến hơn là một gốc phát quang hóa học. Trình tự nucleotide của mẫu dò DNA bổ sung cho trình tự mRNA của gen và do đó được gọi là DNA bổ sung (cDNA) (complementary DNA). Theo thông lệ, người ta sử dụng cDNA được đánh dấu (chứ không phải mRNA được đánh dấu) để dò các thấm Northern vì các phân tử DNA ổn định hơn nhiều và dễ dàng thao tác và nhân lên (thường trong các plasmid của vi khuẩn) hơn các phân tử mRNA. Thấm Northern cung cấp thông tin về lượng (ước tính bằng cường độ tín hiệu trên chụp tự ghi phóng xạ) và kích thước (ước tính bằng vị trí của tín hiệu trên gel so với các tiêu chuẩn được điện di đồng thời) của mRNA cụ thể. Mặc dù kỹ thuật thấm Northern đại diện cho một phương pháp linh hoạt và đơn giản để phân tích mRNA, nó có những nhược điểm lớn, và hiện nay nó đã được thay thế bằng các kỹ thuật nhạy hơn và tốn ít thời gian hơn sẽ được thảo luận sau.
Một trong những phương pháp nhạy nhất để phát hiện và định lượng mRNA hiện có là kỹ thuật PCR phiên mã ngược định lượng (qRT-PCR) (quantitative reverse transcriptase-PCR). Kỹ thuật này kết hợp chức năng độc đáo của enzyme phiên mã ngược với sức mạnh của PCR. qRT-PCR cực kỳ nhạy, cho phép phân tích biểu hiện gen từ lượng RNA rất nhỏ. Hơn nữa, kỹ thuật này có thể được áp dụng cho một số lượng lớn mẫu hoặc nhiều gen (đa mồi) trong cùng một thí nghiệm. Hai tính năng quan trọng này mang lại cho kỹ thuật này một mức độ linh hoạt không có ở các phương pháp truyền thống hơn, chẳng hạn như thấm Northern hoặc phân tích lai hóa trong dung dịch. Bước đầu tiên trong phân tích qRT-PCR là sản xuất DNA bổ sung (cDNA) cho mRNA quan tâm. Điều này được thực hiện bằng cách sử dụng các enzyme có hoạt động DNA polymerase phụ thuộc RNA thuộc nhóm enzyme phiên mã ngược (RT) (ví dụ, virus bệnh bạch cầu chuột Moloney , enzyme phiên mã ngược virus u tủy bào gia cầm), , một DNA polymerase phụ thuộc RNA). Enzyme RT, với sự hiện diện của một mồi thích hợp, sẽ tổng hợp DNA bổ sung cho RNA. Bước thứ hai trong phân tích qRT-PCR là khuếch đại DNA mục tiêu, trong trường hợp này là cDNA được tổng hợp bởi enzyme RT. Tính đặc hiệu của việc khuếch đại được xác định bởi tính đặc hiệu của cặp mồi được sử dụng cho việc khuếch đại PCR. Để xác định tính xác thực của quá trình khuếch đại, danh tính của DNA được khuếch đại có thể được phân tích bằng điện di, lai với mẫu dò RNA hoặc DNA, cắt bằng (các) enzyme giới hạn có thông tin, hoặc được giải trình tự DNA trực tiếp.
Trong khi việc phát hiện một mRNA cụ thể bằng kỹ thuật này tương đối đơn giản, việc định lượng chính xác mRNA trong một mẫu nhất định lại phức tạp hơn. Bởi vì việc sản xuất DNA bằng PCR liên quan đến sự gia tăng theo cấp số nhân của lượng DNA được tổng hợp, những khác biệt tương đối nhỏ trong bất kỳ biến số nào kiểm soát tốc độ khuếch đại sẽ gây ra sự khác biệt rõ rệt về sản lượng của DNA được khuếch đại. Ngoài lượng DNA khuôn, các biến số có thể ảnh hưởng đến sản lượng của PCR bao gồm nồng độ của enzyme polymerase, magiê, nucleotide (dNTPs), và các mồi. Các chi tiết cụ thể của quy trình khuếch đại, bao gồm độ dài chu kỳ, số chu kỳ, nhiệt độ bắt cặp, kéo dài, và biến tính, cũng ảnh hưởng đến sản lượng DNA. Do có vô số biến số liên quan, RT-PCR thông thường không phù hợp để thực hiện phân tích định lượng mRNA. Để khắc phục những cạm bẫy này, các chiến lược thay thế đã được phát triển. Một kỹ thuật để xác định nồng độ của một mRNA cụ thể trong một mẫu sinh học là một sửa đổi của kỹ thuật PCR cơ bản được gọi là RT-PCR cạnh tranh (competitive RT-PCR). Phương pháp này dựa trên việc đồng khuếch đại một DNA đột biến có thể được khuếch đại với cùng một cặp mồi đang được sử dụng cho DNA mục tiêu. DNA đột biến được thiết kế theo cách mà nó có thể được phân biệt với DNA quan tâm bằng kích thước hoặc bằng cách bao gồm một vị trí enzyme giới hạn duy nhất cho DNA đột biến. Việc thêm một lượng tương đương DNA đột biến này vào tất cả các ống phản ứng PCR đóng vai trò là một đối chứng nội bộ cho hiệu quả của quá trình PCR, và sản lượng của DNA đột biến trong các ống khác nhau có thể được sử dụng để cân bằng sản lượng của DNA bằng PCR. Điều quan trọng là phải đảm bảo để định lượng chính xác DNA quan tâm rằng nồng độ của khuôn đột biến và khuôn mục tiêu phải gần như tương đương. Vì việc sử dụng DNA đột biến để chuẩn hóa không tính đến sự thay đổi về hiệu quả của enzyme RT, một biến thể của phương pháp ban đầu đã được phát triển. Trong sửa đổi này, RNA đột biến cạnh tranh được phiên mã từ một vector biểu hiện RNA được thiết kế phù hợp được thay thế cho DNA đột biến trong phản ứng trước khi bắt đầu tổng hợp cDNA. RT-PCR cạnh tranh có thể được sử dụng để phát hiện những thay đổi cỡ hai đến ba lần của ngay cả những loài mRNA rất hiếm. Nhược điểm chính của phương pháp này là xu hướng cho kết quả không chính xác do sự nhiễm bẩn của các mẫu với mRNA quan tâm. Về lý thuyết, vì kỹ thuật này dựa trên PCR, sự nhiễm bẩn bởi dù chỉ một phân tử mRNA quan tâm cũng có thể làm mất giá trị của kết quả. Do đó, sự chú ý tỉ mỉ đến kỹ thuật và thiết lập phòng thí nghiệm là điều cần thiết cho việc áp dụng thành công kỹ thuật này.
Nói chung, hai loại phương pháp được sử dụng để phát hiện và định lượng các sản phẩm PCR: các phép đo sản phẩm “điểm cuối” và các kỹ thuật “thời gian thực” mới hơn. Các phép xác định điểm cuối (ví dụ, kỹ thuật RT-PCR cạnh tranh được mô tả trước đó) phân tích phản ứng sau khi nó hoàn thành, trong khi các phép xác định thời gian thực được thực hiện trong quá trình khuếch đại. Nói chung, phương pháp thời gian thực chính xác hơn và hiện là phương pháp được ưa chuộng. Những tiến bộ trong công nghệ phát hiện huỳnh quang đã giúp việc sử dụng đo lường thời gian thực trở nên khả thi cho việc sử dụng thường quy trong phòng thí nghiệm. Một trong những kỹ thuật phổ biến tận dụng các phép đo thời gian thực là xét nghiệm TaqMan (5′ nuclease huỳnh quang) (fluorescent 5′ nuclease assay) (Hình 2.5). Thiết kế độc đáo của các mẫu dò TaqMan, kết hợp với hoạt động 5′ nuclease của enzyme PCR (Taq polymerase), cho phép phát hiện trực tiếp sản phẩm PCR bằng cách giải phóng một chất báo cáo huỳnh quang trong quá trình khuếch đại PCR bằng cách sử dụng các máy được thiết kế đặc biệt (ABI Prism 5700/7700). Mẫu dò TaqMan bao gồm một oligonucleotide được tổng hợp với một thuốc nhuộm báo cáo 5′ (ví dụ, FAM; 6-carboxy-fluorescein) và một thuốc nhuộm dập tắt 3′ xuôi dòng (ví dụ, TAMRA; 6-carboxy-tetramethyl-rhodamine). Khi mẫu dò còn nguyên vẹn, sự gần gũi của thuốc nhuộm báo cáo với thuốc nhuộm dập tắt dẫn đến sự triệt tiêu huỳnh quang của chất báo cáo, chủ yếu bằng cách truyền năng lượng kiểu Forster. Trong quá trình PCR, các mồi xuôi và ngược lai với một trình tự cụ thể của DNA mục tiêu. Mẫu dò TaqMan lai với một trình tự mục tiêu trong sản phẩm PCR. Enzyme Taq polymerase, do hoạt động 5′-3′ exonuclease của nó, sau đó cắt mẫu dò TaqMan. Thuốc nhuộm báo cáo và thuốc nhuộm dập tắt được tách ra bởi sự cắt, dẫn đến sự gia tăng huỳnh quang của thuốc nhuộm báo cáo như một hệ quả trực tiếp của việc khuếch đại mục tiêu trong quá trình PCR. Quá trình này xảy ra trong mọi chu kỳ và không cản trở sự tích lũy theo cấp số nhân của sản phẩm. Cả mồi và mẫu dò đều phải lai với mục tiêu để quá trình khuếch đại và cắt xảy ra. Tín hiệu huỳnh quang chỉ được tạo ra nếu trình tự mục tiêu cho mẫu dò được khuếch đại trong quá trình PCR. Do những yêu cầu nghiêm ngặt này, sự khuếch đại không đặc hiệu không được phát hiện. Việc phát hiện huỳnh quang diễn ra thông qua các đường cáp quang được đặt phía trên các nắp ống không bị biến dạng quang học. Dữ liệu định lượng được lấy từ việc xác định chu kỳ mà tại đó tín hiệu sản phẩm khuếch đại vượt qua một ngưỡng phát hiện được đặt trước. Số chu kỳ này tỷ lệ thuận với lượng vật liệu ban đầu, do đó cho phép đo lường mức độ mRNA cụ thể trong mẫu. Một máy thay thế (Light Cycler) cũng sử dụng các mẫu dò thủy phân fluorogenic hoặc lai hóa fluorogenic để định lượng theo cách tương tự như hệ thống ABI.
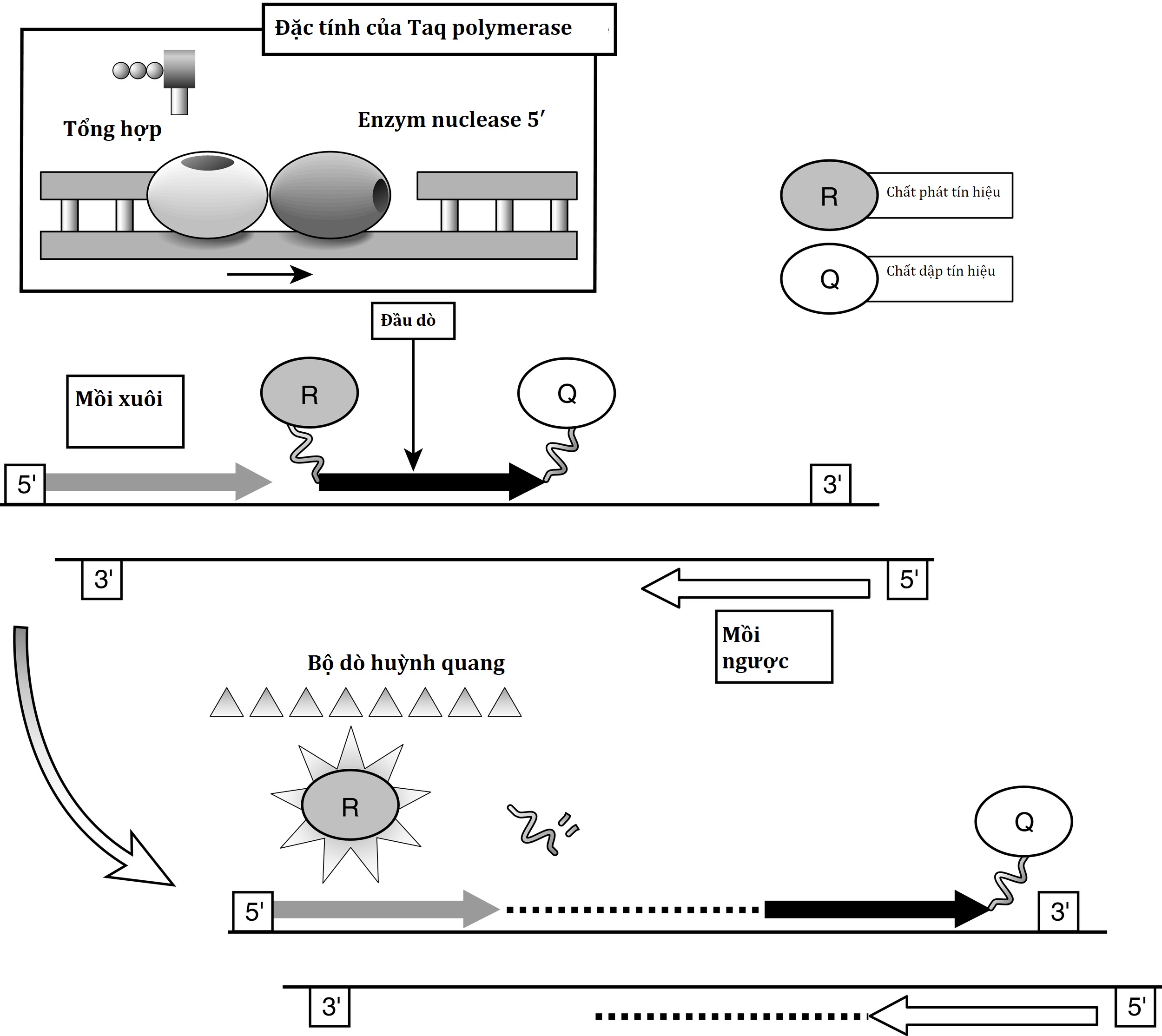
Hình 2.5 Xét nghiệm 5′ nuclease huỳnh quang (TaqMan). Ba oligonucleotide tổng hợp được sử dụng trong xét nghiệm 5′ nuclease huỳnh quang. Hai oligonucleotide hoạt động như mồi “xuôi” và “ngược” trong một quy trình khuếch đại phản ứng chuỗi polymerase (PCR) thông thường. Oligonucleotide thứ ba, được gọi là mẫu dò TaqMan, bao gồm một oligonucleotide được tổng hợp với một thuốc nhuộm báo cáo 5′ (ví dụ, FAM; 6-carboxy-fluorescein) và một thuốc nhuộm dập tắt 3′ xuôi dòng (ví dụ, TAMRA; 6-carboxy-tetramethyl-rhodamine). Khi mẫu dò còn nguyên vẹn, sự gần gũi của thuốc nhuộm báo cáo với thuốc nhuộm dập tắt dẫn đến sự triệt tiêu huỳnh quang của chất báo cáo, chủ yếu bằng cách truyền năng lượng kiểu Forster. Trong quá trình PCR, các mồi xuôi và ngược lai với một trình tự cụ thể của DNA mục tiêu. Mẫu dò TaqMan lai với một trình tự mục tiêu trong sản phẩm PCR. Enzyme Taq polymerase, do hoạt động 5′-3′ exonuclease của nó, sau đó cắt mẫu dò TaqMan. Thuốc nhuộm báo cáo và thuốc nhuộm dập tắt được tách ra bởi sự cắt, dẫn đến sự gia tăng huỳnh quang của thuốc nhuộm báo cáo như một hệ quả trực tiếp của việc khuếch đại mục tiêu trong quá trình PCR. Cả mồi và mẫu dò đều phải lai với mục tiêu để quá trình khuếch đại và cắt xảy ra. Do đó, tín hiệu huỳnh quang chỉ được tạo ra nếu trình tự mục tiêu cho mẫu dò được khuếch đại trong quá trình PCR. Việc phát hiện huỳnh quang diễn ra thông qua các đường cáp quang được đặt phía trên nắp của các giếng phản ứng. Hình nhỏ: Hai chức năng riêng biệt của enzyme Taq polymerase: hoạt động polymerase tổng hợp 5′-3′ và hoạt động exonuclease phụ thuộc polymerase 6′-3′.
MicroRNA
Một trong những tiến bộ đáng kể vào đầu những năm 2000 trong lĩnh vực sinh học RNA là việc phát hiện ra các RNA không mã hóa nhỏ (20-30 nucleotide). Nói chung, có hai loại RNA không mã hóa nhỏ: microRNA (miRNA) và RNA can thiệp nhỏ (siRNA) (small interfering RNA). miRNA là các sản phẩm biểu hiện của bộ gen của chính sinh vật, trong khi siRNA được tổng hợp trong tế bào từ RNA sợi đôi ngoại lai (ví dụ, từ virus hoặc transposon hoặc từ DNA tổng hợp được đưa vào tế bào để nghiên cứu chức năng của một gen/quá trình cụ thể). Ngoài ra, có sự khác biệt trong quá trình sinh tổng hợp của hai loại RNA nucleotide nhỏ này. Bất chấp những khác biệt này, hiệu ứng sinh học tổng thể của các RNA nucleotide nhỏ này là ức chế dịch mã hoặc phân hủy mục tiêu và làm câm lặng gen bằng cách liên kết với các trình tự bổ sung trên vùng không dịch mã 3′ của mRNA mục tiêu; sự điều hòa dương tính của biểu hiện gen thông qua một cơ chế như vậy là rất hiếm. Sự phức tạp của hiện tượng này tăng lên bởi thực tế là trong bối cảnh đặc hiệu của tế bào hoặc mô, một miRNA duy nhất có thể nhắm vào nhiều RNA và nhiều hơn một miRNA có thể nhận ra cùng một mRNA mục tiêu để khuếch đại và tăng cường sự ức chế dịch mã của gen mục tiêu. Ước tính rằng hiện tượng này có mặt ở một số loại tế bào và bộ gen người mã hóa cho hơn 1000 miRNA có thể nhắm vào 60% đến 70% các gen của động vật có vú. Các sự kiện qua trung gian miRNA đã được liên quan đến việc điều hòa sự tăng trưởng và biệt hóa tế bào, sự phát triển tế bào, apoptosis, và các quá trình tế bào khác. Cho đến nay, tác động lớn của việc phát hiện miRNA đã nằm trong các lĩnh vực sinh học phát triển, sự hình thành cơ quan, và ung thư. miRNA và các sự kiện liên quan đến miRNA (ví dụ, các protein liên quan đến quá trình xử lý miRNA) đã được liên quan trực tiếp đến chỉ một số ít các rối loạn nội tiết không phải ung thư (ví dụ, hội chứng diGeorge và chậm phát triển tâm thần liên kết X). Dự đoán rằng khi chúng ta tìm hiểu thêm về sinh học cơ bản của quá trình này, các RNA không mã hóa nucleotide nhỏ sẽ được liên quan đến cơ chế bệnh sinh của một phổ rộng hơn các bệnh nội tiết.
PHÁT HIỆN ĐỘT BIẾN TRONG GEN
Những thay đổi trong tổ chức cấu trúc của một gen ảnh hưởng đến chức năng của nó bao gồm mất đoạn, chèn đoạn, hoặc chuyển vị các đoạn DNA tương đối lớn, hoặc thường xuyên hơn là các thay thế base đơn trong các vùng chức năng quan trọng. Giải trình tự thông lượng cao (High throughput sequencing) hay giải trình tự thế hệ mới (NGS) (next-generation sequencing) đã cách mạng hóa việc xác định các đột biến trong gen.
Các phương pháp trực tiếp
Giải trình tự DNA (DNA sequencing) là tiêu chuẩn vàng hiện tại để có được bằng chứng rõ ràng về một đột biến điểm. Tuy nhiên, giải trình tự DNA có những hạn chế và nhược điểm của nó. Một vấn đề liên quan đến lâm sàng là các phương pháp giải trình tự DNA hiện tại không phát hiện được tất cả các đột biến một cách đáng tin cậy và nhất quán. Ví dụ, trong nhiều trường hợp đột biến chỉ ảnh hưởng đến một alen (dị hợp tử), chiều cao của các đỉnh base trên biểu đồ huỳnh quang tương ứng với alen kiểu dại và alen đột biến không phải lúc nào cũng có mặt theo tỷ lệ dự đoán (1:1). Điều này giới hạn sức mạnh phân biệt của các giao thức máy tính “gọi base” và dẫn đến việc gán trình tự DNA không nhất quán hoặc sai lầm cho các alen riêng lẻ. Do hạn chế này, các phòng thí nghiệm lâm sàng thường xác định trình tự DNA của cả hai alen để cung cấp xác nhận độc lập về sự vắng mặt/hiện diện của một đột biến giả định. Giải trình tự DNA có thể tốn nhiều công sức và chi phí, mặc dù những tiến bộ trong pyrosequencing (sẽ thảo luận sau), ví dụ, đã làm cho nó trở nên dễ dàng và rẻ hơn về mặt kỹ thuật.
Mặc dù các trình tự DNA đầu tiên được xác định bằng một phương pháp cắt DNA về mặt hóa học tại mỗi trong bốn nucleotide, phương pháp enzyme hay dideoxy (enzymatic or dideoxy method) do Sanger và các đồng nghiệp phát triển năm 1977 đã trở thành phương pháp được sử dụng phổ biến nhất cho các mục đích thông thường (Hình 2.6). Phương pháp này sử dụng enzyme DNA polymerase để tổng hợp một bản sao bổ sung của DNA sợi đơn (“khuôn”) có trình tự đang được xác định. DNA sợi đơn có thể được thu nhận trực tiếp từ các vector virus hoặc plasmid hỗ trợ tạo ra DNA sợi đơn hoặc bằng cách biến tính một phần DNA sợi đôi bằng cách xử lý với kiềm hoặc nhiệt. Enzyme DNA polymerase không thể tự bắt đầu tổng hợp một chuỗi DNA mới mà chỉ có thể kéo dài một đoạn DNA. Do đó, yêu cầu thứ hai cho phương pháp giải trình tự dideoxy là sự hiện diện của một “mồi”. Mồi là một oligonucleotide tổng hợp, dài từ 15 đến 30 base, có trình tự bổ sung với trình tự của đoạn tương ứng ngắn của khuôn DNA sợi đơn. Phương pháp dideoxy khai thác quan sát rằng DNA polymerase có thể sử dụng cả dNTP và 2′,3′-dideoxynucleoside triphosphates (ddNTPs) làm cơ chất trong quá trình kéo dài mồi. Trong khi DNA polymerase có thể sử dụng dNTP để tiếp tục tổng hợp sợi DNA bổ sung, chuỗi không thể kéo dài thêm sau khi thêm ddNTP đầu tiên, vì ddNTP thiếu nhóm 3′-hydroxyl quan trọng. Để xác định nucleotide ở cuối chuỗi, bốn phản ứng được thực hiện cho mỗi phân tích trình tự, với chỉ một trong bốn ddNTP có thể có được bao gồm trong bất kỳ một phản ứng nào. Tỷ lệ ddNTP và dNTP trong mỗi phản ứng được điều chỉnh sao cho các kết thúc chuỗi này xảy ra tại mỗi vị trí trong khuôn nơi nucleotide đó xuất hiện. Để cho phép phát hiện bằng chụp tự ghi phóng xạ, DNA mới tổng hợp được đánh dấu, thường bằng cách bao gồm trong hỗn hợp phản ứng dATP được đánh dấu phóng xạ (cho các phương pháp thủ công cũ hơn) hoặc, phổ biến nhất hiện nay, là các chất kết thúc nhuộm huỳnh quang trong hỗn hợp phản ứng (hiện đang được sử dụng trong các kỹ thuật tự động). Việc phân tách các sợi DNA mới được tổng hợp theo cách thủ công được thực hiện thông qua điện di polyacrylamide biến tính có độ phân giải cao hoặc với điện di mao quản trong các máy giải trình tự tự động. Các phương pháp phát hiện huỳnh quang đã cho phép tự động hóa và tăng cường thông lượng. Trong điện di mao quản, các phân tử DNA được thúc đẩy di chuyển qua một polymer nhớt bởi một điện trường cao để được phân tách dựa trên điện tích và kích thước. Mặc dù kỹ thuật này dựa trên cùng nguyên tắc được sử dụng trong điện di trên bản gel, việc phân tách được thực hiện trong các mao quản thủy tinh riêng lẻ thay vì các bản gel, tạo điều kiện thuận lợi cho việc nạp mẫu và các khía cạnh khác của tự động hóa. Trong khi các phương pháp thủ công cho phép phát hiện khoảng 300 nucleotide thông tin trình tự với một bộ phản ứng giải trình tự, các phương pháp tự động sử dụng thuốc nhuộm huỳnh quang và công nghệ laser có thể phân tích 7500 base trở lên cho mỗi phản ứng. Để giải trình tự các đoạn DNA lớn hơn, cần phải chia đoạn DNA lớn thành các đoạn nhỏ hơn có thể được giải trình tự riêng lẻ. Ngoài ra, các mồi giải trình tự bổ sung có thể được chọn gần cuối kết quả giải trình tự trước đó, cho phép điểm bắt đầu của dữ liệu trình tự mới được di chuyển dần dần dọc theo đoạn DNA lớn hơn.
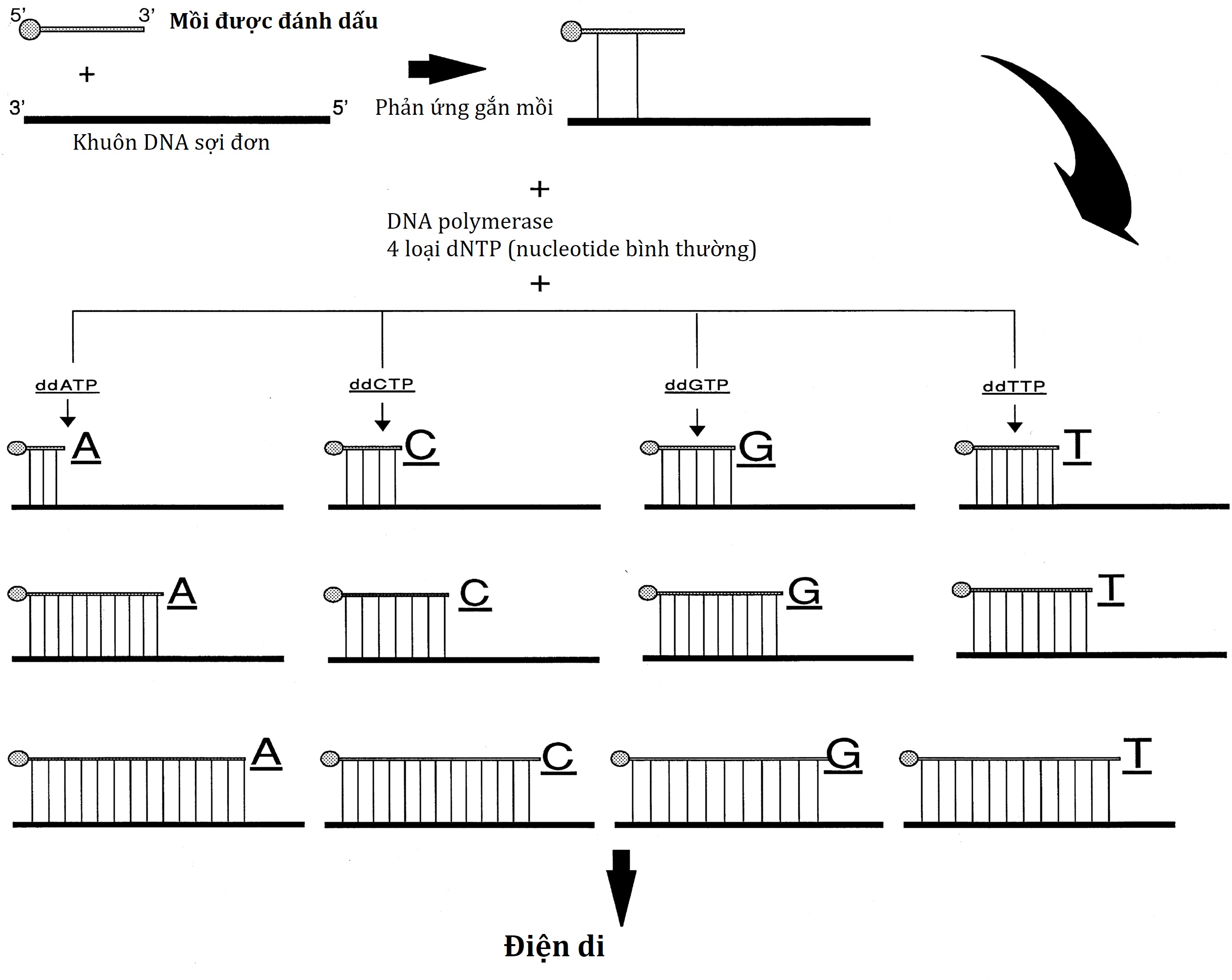
Hình 2.6 Giải trình tự DNA bằng phương pháp dideoxy (Sanger). Một mồi oligonucleotide được đánh dấu đầu 5′ có trình tự bổ sung với DNA cần được giải trình tự (khuôn DNA) được bắt cặp vào một sợi đơn của khuôn DNA. Mồi này được kéo dài bằng quá trình tổng hợp DNA được khởi đầu bằng việc thêm enzyme DNA polymerase với sự hiện diện của bốn 2′-deoxynucleoside triphosphate (dNTPs) và một trong các 2′,3′-dideoxynucleoside triphosphate (ddNTPs); bốn ống phản ứng như vậy được lắp ráp để sử dụng tất cả bốn ddNTP. Enzyme DNA polymerase sẽ kéo dài mồi bằng cách sử dụng các dNTP và ddNTP riêng lẻ có trong ống phản ứng cụ thể đó. Bởi vì ddNTP không có nhóm hydroxyl 3′, không thể kéo dài chuỗi khi một gốc như vậy được thêm vào chuỗi. Do đó, mỗi ống phản ứng sẽ chứa các chuỗi bị kết thúc sớm tại vị trí xuất hiện của ddNTP cụ thể có trong ống phản ứng. Nồng độ của các dNTP và ddNTP riêng lẻ có trong các ống phản ứng được điều chỉnh sao cho việc kết thúc chuỗi diễn ra tại mọi lần xuất hiện của ddNTP. Sau phản ứng kéo dài-kết thúc chuỗi, các sợi DNA được tổng hợp được phân tách theo kích thước bằng điện di trên gel acrylamide và các dải được hiển thị bằng chụp tự ghi phóng xạ.
Một trong những tiến bộ công nghệ tinh túy là sự ra đời của các phương pháp dựa trên vi mảng (microarray) (microarray) để phát hiện và phân tích các acid nucleic. Các vi mảng chứa hàng ngàn oligonucleotide được lắng đọng hoặc tổng hợp tại chỗ trên một giá đỡ rắn, thường là một phiến kính được phủ hoặc một màng. Trong kỹ thuật này, một thiết bị robot được sử dụng để in các trình tự DNA lên giá đỡ rắn. Các mẫu dò DNA được cố định trên phiến vi mảng dưới dạng các đốm có thể là cDNA được nhân bản hoặc các đoạn gen (thẻ trình tự biểu hiện [ESTs]), hoặc các oligonucleotide tương ứng với các gen đã biết hoặc các khung đọc mở giả định. Các mảng được lai với các mục tiêu huỳnh quang được chuẩn bị từ RNA chiết xuất từ mô/tế bào quan tâm; RNA được đánh dấu bằng các thẻ huỳnh quang, chẳng hạn như Cy3 và Cy5. Mô hình thí nghiệm vi mảng nguyên mẫu bao gồm việc so sánh sự phong phú của mRNA trong hai mẫu khác nhau. Một mục tiêu huỳnh quang được chuẩn bị từ mRNA đối chứng và mục tiêu thứ hai với một nhãn huỳnh quang khác được chuẩn bị từ mRNA được phân lập từ các tế bào hoặc mô được xử lý đang được điều tra. Cả hai mục tiêu được trộn lẫn và lai với phiến vi mảng, dẫn đến việc các trình tự gen mục tiêu lai với các trình tự bổ sung của chúng trên phiến vi mảng. Vi mảng sau đó được kích thích bằng laser, và cường độ huỳnh quang của mỗi đốm được xác định với cường độ tương đối của hai tín hiệu màu trên các đốm riêng lẻ tỷ lệ thuận với lượng bản sao mRNA cụ thể trong mỗi mẫu (Hình 2.7). Phân tích dữ liệu cường độ huỳnh quang mang lại một ước tính về mức độ biểu hiện tương đối của các gen trong mẫu thử và mẫu đối chứng. Vi mảng cho phép các nhà điều tra cá nhân thực hiện các phân tích quy mô lớn trên các sinh vật mô hình và tùy chỉnh các mảng cho các ứng dụng bộ gen đặc biệt.
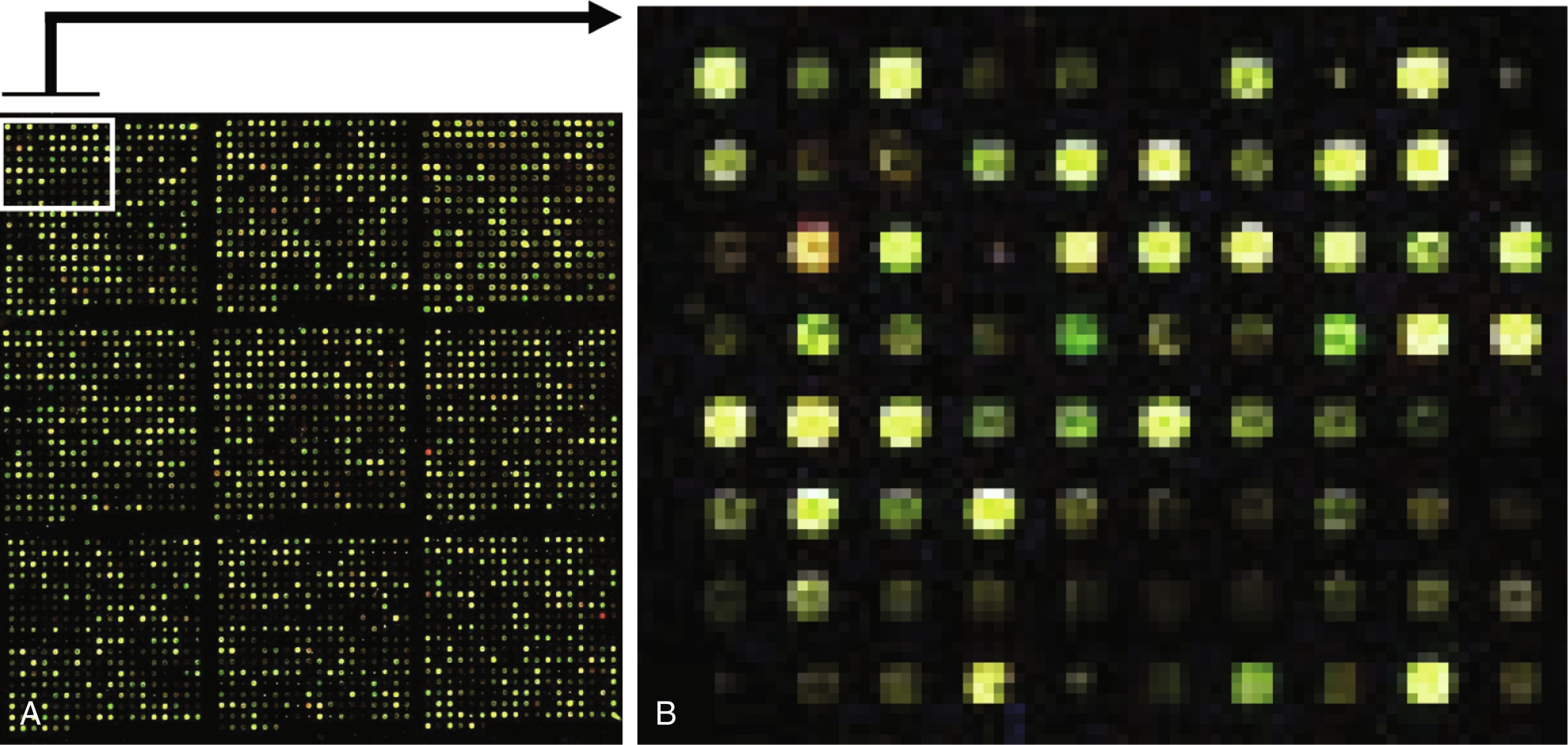
Hình 2.7 A, Vi mảng DNA bổ sung (cDNA), các mục tiêu cDNA được đánh dấu huỳnh quang, tăng sản thượng thận dạng nốt lớn hai bên không phụ thuộc ACTH (Cy3), và tăng sản phụ thuộc ACTH (Cy5) đã được lai với các phiến kính chứa các gen liên quan đến quá trình sinh ung thư. Sau khi kích hoạt các thẻ huỳnh quang bằng laser, các tín hiệu huỳnh quang từ mỗi “đốm” DNA được ghi lại và được phân tích. B, Chế độ xem phóng đại của nền tảng vi mảng hiển thị các tín hiệu huỳnh quang; màu xanh lá cây (Cy3) và màu đỏ (Cy5) với màu vàng đại diện cho sự chồng chéo của hai màu này.
Phương pháp được lựa chọn để lập hồ sơ biểu hiện toàn cầu phụ thuộc vào một số yếu tố, bao gồm các khía cạnh kỹ thuật, lao động, giá cả, thời gian và công sức liên quan, và quan trọng nhất là loại thông tin được tìm kiếm. Những tiến bộ kỹ thuật trong việc phát triển các mảng biểu hiện, sự phong phú và sẵn có trên thị trường của chúng, và tốc độ tương đối mà phân tích có thể được thực hiện là tất cả các yếu tố làm cho các mảng trở nên hữu ích hơn trong các ứng dụng thông thường. Ngoài ra, nội dung của mảng hiện có thể được tùy chỉnh dễ dàng để bao phủ từ các cụm gen và các con đường quan tâm đến toàn bộ bộ gen: một số nghiên cứu kiểm tra một loạt các bản sao đặc hiệu cho mô hoặc các gen được biết là có liên quan đến bệnh lý cụ thể; những nghiên cứu khác trực tiếp sử dụng các mảng bao phủ toàn bộ bộ gen. Một yếu tố khác cần được xem xét trước khi bắt tay vào bất kỳ phương pháp thông lượng cao nào là liệu các mẫu riêng lẻ hay gộp sẽ được điều tra. Một loạt các mẫu gộp làm giảm giá cả, thời gian bỏ ra, và số lượng các thí nghiệm xuống mức phải chăng nhất. Tuy nhiên, việc điều tra các mẫu riêng lẻ là quan trọng để xác định các tỷ lệ biểu hiện duy nhất trong một loại mô hoặc tế bào nhất định. Có những hạn chế của các kỹ thuật dựa trên vi mảng; ví dụ, tương tự như các phương pháp giải trình tự DNA trực tiếp, các phương pháp dựa trên vi mảng cũng gặp phải bất lợi là không thể phát hiện các đột biến dị hợp tử một cách đáng tin cậy và nhất quán. Hơn nữa, vi mảng không thể được sử dụng để phát hiện việc chèn nhiều nucleotide mà không làm tăng theo cấp số nhân số lượng oligonucleotide phải được cố định trên các phiến kính.
Một kỹ thuật đáng tin cậy hơn trong việc xác định đột biến là pyrosequencing, dựa trên việc theo dõi thời gian thực bằng enzyme quá trình tổng hợp DNA bằng phát quang sinh học (Hình 2.8). Pyrosequencing được thực hiện bằng cách thêm các dNTP riêng lẻ, theo một thứ tự phân phối được xác định trước, sao cho chuỗi nucleotide non được kéo dài thêm một gốc nucleotide cho mỗi sự kiện phân phối. Việc phát hiện trình tự nucleotide được thực hiện thông qua một chuỗi các phản ứng enzyme liên quan đến hoạt động của DNA polymerase, apyrase, ATP sulfurylase, và luciferase, tương ứng, cho phép kết hợp nucleotide bổ sung, phân hủy dNTP không sử dụng, tạo ra cơ chất luciferase từ pyrophosphate và adenosine 5′-phosphosulfate, và phát ra ánh sáng từ việc chuyển đổi luciferin thành oxyluciferin do ATP điều khiển. Việc kết hợp một nucleotide cụ thể được hiển thị đồ thị dưới dạng một biểu đồ ghi lại sự kiện phân phối nucleotide so với cường độ ánh sáng phát ra. Chuỗi phản ứng enzyme này có tính định lượng, ở chỗ cường độ ánh sáng tăng lên khi kết hợp nhiều nucleotide.
Pyrosequencing, được giới thiệu vào đầu những năm 2000, đã cung cấp nền tảng cho sự bùng nổ của các kỹ thuật mới được gọi chung là NGS thông lượng cao hay giải trình tự song song hàng loạt (massively parallel sequencing). NGS cung cấp độ dài đọc dài hơn và giá mỗi base giải trình tự rẻ hơn so với giải trình tự Sanger. NGS dựa trên việc tách rời phản ứng enzyme xác định nucleotide truyền thống và việc chụp ảnh và thực hiện điều đó một cách ngày càng nhanh hơn cho phép dung lượng gần như không giới hạn.
Những khám phá đầu tiên về đột biến gen gây bệnh nội tiết khai thác NGS đã được công bố vào năm 2011. Hiện tại, nhiều hệ thống tương tự đang được sử dụng cho NGS. Ví dụ, quy trình làm việc của Illumina bao gồm bốn bước cơ bản: bước đầu tiên bao gồm việc phân mảnh ngẫu nhiên mẫu DNA (hoặc cDNA), sau đó là gắn adapter 5′ và 3′. Các trình tự adapter ngắn này bao gồm các vị trí liên kết, chỉ số (cần thiết để thực hiện các phản ứng đa mồi), và các đoạn bổ sung cho các oligo được cố định trên một flow cell. Các đoạn được gắn adapter sau đó được khuếch đại bằng PCR và tinh sạch bằng gel để tạo ra một “thư viện”. Sau đó, thư viện được tạo ra được nạp vào flow cell, nơi các đoạn của nó được bắt giữ trên các oligo bổ sung với các adapter của thư viện, được cố định trên một giá đỡ rắn ở nồng độ loãng. Mỗi đoạn sau đó được khuếch đại thêm thành các cụm nhân bản riêng biệt thông qua khuếch đại cầu. “Cầu” được hình thành bằng cách bắt cặp của trình tự adapter đầu cuối, vẫn còn tự do ở đầu kia, chưa được bắt giữ của mỗi đoạn đơn, uốn cong đến oligo cố định bổ sung liền kề. Một polymerase sau đó tạo ra một sợi bổ sung. Sau khi biến tính, cả hai sợi lại liên kết, bắt cặp với các mồi bổ sung khác được neo vào sàn của đĩa. Bước này được lặp lại một số lần, tạo ra hàng triệu bản sao của mỗi đoạn. Khi “quá trình tạo cụm” này hoàn tất, các khuôn đã sẵn sàng để “giải trình tự”. Ở đây cũng vậy, theo pyrosequencing thông thường, phương pháp dựa trên chất kết thúc có thể đảo ngược phát hiện các base đơn khi chúng được kết hợp vào các sợi khuôn DNA, tạo ra sự phát xạ ánh sáng dưới sự kích thích bằng laser. Mỗi cụm của các đoạn riêng biệt được giải trình tự thông qua việc chụp các hình ảnh có độ phân giải cao phản ánh việc thêm từng base của nucleotide. Trái ngược với pyrosequencing thông thường, tất cả bốn dNTP được gắn chất kết thúc có thể đảo ngược đều có mặt trong mỗi chu kỳ giải trình tự, mỗi loại được đánh dấu bằng một thuốc nhuộm huỳnh quang khác nhau. Sự cạnh tranh tự nhiên giảm thiểu sai lệch kết hợp, làm giảm tỷ lệ lỗi thô. Cuối cùng, một chương trình máy tính tinh vi sẽ sắp xếp các lần đọc trình tự mới được xác định vào một trình tự bộ gen tham chiếu, được nhóm lại, trong trường hợp các phản ứng đa mồi, bằng các đoạn chỉ số cho mỗi nguồn DNA khác nhau. Số lượng cực kỳ cao các trình tự giống nhau đảm bảo tính chính xác của kết quả thu được và loại trừ những kết quả chỉ được biểu hiện lẻ tẻ. Sự gia tăng đáng kể về sản lượng dữ liệu này, cùng với việc giảm dần chi phí giải trình tự, đã mở đường cho “y học cá nhân hóa”, trong đó bộ gen của mỗi cá nhân có thể dễ dàng thu được và so sánh với các trình tự bộ gen bình thường để phát hiện các đột biến liên quan đến bệnh, đã biết hoặc mới, có thể có.
Các hệ thống dựa trên chất bán dẫn và công nghệ nano hiện đang được sử dụng trong các nỗ lực giải trình tự hàng loạt và hứa hẹn một cách thậm chí còn rẻ hơn và nhanh hơn để xác định các đột biến và các bất thường khác của bộ gen người.
Một yêu cầu của tất cả các phương pháp sàng lọc thông lượng cao là xác nhận các phát hiện (mức độ biểu hiện của một gen/trình tự nhất định) bằng các phương pháp độc lập khác. Một nhóm gen được chọn thường được kiểm tra; các gen này được chọn từ một loạt các trình tự đã được phân tích hoặc vì chúng được phát hiện có những thay đổi đáng kể hoặc vì sự quan tâm đặc biệt đến biểu hiện của chúng trong mô được nghiên cứu hoặc mối quan hệ đã được xác định trước đó của chúng với bệnh lý hoặc giai đoạn phát triển. Quá trình xác nhận cố gắng hỗ trợ các phát hiện trên ba cấp độ khác nhau: (1) độ tin cậy của thí nghiệm thông lượng cao (cho mục đích này, các mẫu tương tự được kiểm tra bởi các vi mảng được sử dụng); (2) tính trung thực của các quan sát nói chung (để đạt được điều đó, số lượng mẫu lớn hơn được kiểm tra, việc đánh giá bằng các phương pháp thông lượng cao thường không thể chi trả được về giá cả hoặc lao động); và (3) xác minh những thay đổi biểu hiện ở cấp độ protein. Một kỹ thuật xác nhận thường được sử dụng là qRT-PCR. Để xác minh ở cấp độ protein, hóa mô miễn dịch (IHC) (immunohistochemistry) và thấm Western (Western blot) (Western blot) là hai kỹ thuật được lựa chọn phổ biến nhất. IHC không định lượng nhưng có lợi thế là cho phép quan sát vị trí chính xác của một tín hiệu trong một tế bào (tế bào chất so với nhân) và mô (xác định mô học của mô được nhuộm). Các phương pháp thấm Western hiện đại đòi hỏi một lượng lysate protein nhỏ hơn so với các kỹ thuật cũ và có lợi thế là cung cấp định lượng biểu hiện có độ phân giải cao mà không cần sử dụng phóng xạ.
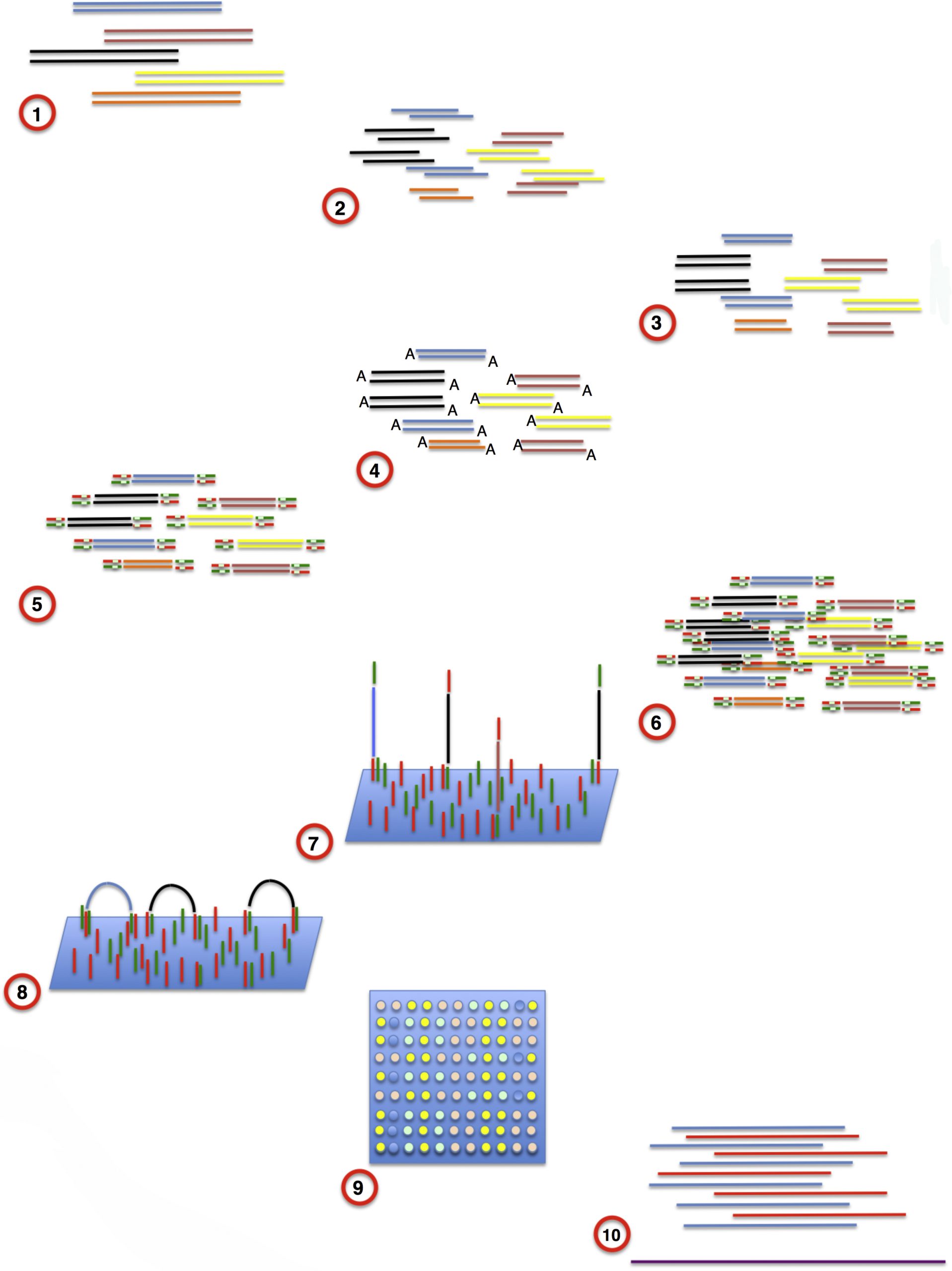
Hình 2.8 Các bước trong pyrosequencing. (1) Chiết xuất DNA. (2) Cắt DNA bộ gen. (3) Sửa chữa đầu đoạn. (4) Gắn Adenine. (5) Gắn Adaptor. (6) Khuếch đại PCR. (7) Gắn đoạn, thông qua các adaptor, vào các đoạn DNA bổ sung được neo trên một bề mặt rắn. (8) Hình thành cụm nhân bản thông qua khuếch đại cầu. (9) Chụp ảnh độ phân giải cao. (10) Sắp xếp nhiều lần đọc vào một bộ gen tham chiếu.
Các phương pháp gián tiếp
Vào giữa những năm 1980, nhu cầu về các hệ thống phân tích đột biến nhanh chóng, thông lượng cao, chính xác và kinh tế đã dẫn đến sự phát triển của một số công nghệ, như một giải pháp thay thế cho việc phân tích bằng giải trình tự trực tiếp, cho phép phát hiện các đột biến đơn trong các đoạn DNA dài (200-600 bp). Tuy nhiên, việc sàng lọc đột biến bằng các phương pháp gián tiếp đã không còn được ưa chuộng vì NGS và giải trình tự Sanger đã trở thành các phương pháp nhanh hơn và rẻ hơn để xác định các đột biến gen. Chúng tôi chỉ đề cập ngắn gọn đến chúng ở đây và vì mục đích lịch sử, bởi vì một số kỹ thuật này vẫn được sử dụng lẻ tẻ, và việc hiểu y văn được xuất bản từ những năm 1980 đòi hỏi kiến thức về các nguyên tắc cơ bản của những kỹ thuật này.
Các phương pháp gián tiếp để xác định đột biến bao gồm cắt sản phẩm PCR bằng endonuclease giới hạn (PCR-RFLP), điện di trên gel gradient biến tính, đa hình hình dạng sợi đơn, dấu vân tay dideoxy, và xét nghiệm di động dị hợp tử. Hầu hết các phương pháp này đều sử dụng PCR để khuếch đại một vùng của DNA, một xử lý vật lý hoặc hóa học của DNA được khuếch đại (ví dụ, bằng cách biến tính hoặc cắt bằng enzyme giới hạn), phân tách các amplicon bằng điện di trên gel biến tính hoặc không biến tính, và hiển thị các sợi trình tự đã phân tách (bằng chụp tự ghi phóng xạ hoặc phát hiện dựa trên huỳnh quang). Các sửa đổi trong một số kỹ thuật này cho phép phân tách và phát hiện đồng thời các đoạn DNA với việc sử dụng các thiết bị phức tạp, chẳng hạn như HPLC và điện di mao quản.
HỆ THỐNG CRISPR-CAS9
Hệ thống CRISPR-Cas9 là một phương pháp mới được phát hiện tương đối đơn giản, nhưng rất hiệu quả để sửa chữa vật lý các đột biến có trên các gen được chọn hoặc để đột biến một trình tự gen mục tiêu theo ý muốn (Hình 2.9). Nó nhanh hơn, rẻ hơn và chính xác hơn các kỹ thuật chỉnh sửa DNA trước đây và có một loạt các ứng dụng tiềm năng cho cả khoa học cơ bản và y học lâm sàng. Đây là một công nghệ độc đáo cho phép các nhà di truyền học và các nhà nghiên cứu y học chỉnh sửa các phần của bộ gen bằng cách loại bỏ, thêm, hoặc thay đổi các đoạn của trình tự DNA. Nó dựa trên một hệ thống giới hạn mới có ở một số vi khuẩn đã tiến hóa để đối phó với các mầm bệnh xâm nhập. Hệ thống này bao gồm hai phân tử chính: một enzyme gọi là Cas9 và một đoạn RNA gọi là RNA dẫn đường (gRNA) (guide RNA). Cas9 hoạt động như một cặp “kéo phân tử” có thể cắt hai sợi DNA tại một vị trí cụ thể trong bộ gen, để các mẩu DNA sau đó có thể được thêm vào hoặc loại bỏ tại cùng một vị trí DNA đó. gRNA là một trình tự RNA ngắn (dài khoảng 20 base) có khả năng liên kết với DNA tại một trình tự được thiết kế trước. gRNA liên kết với một trình tự cụ thể trong DNA, “dẫn đường” cho Cas9 đến phần được chọn của bộ gen nơi nó sẽ cắt DNA. Cơ chế sửa chữa DNA của tế bào sau đó sẽ hoàn thành quá trình sửa chữa dẫn đến việc định hình lại đoạn DNA mục tiêu sẽ không bao gồm trình tự cần loại bỏ hoặc bao gồm một đoạn được lựa chọn.
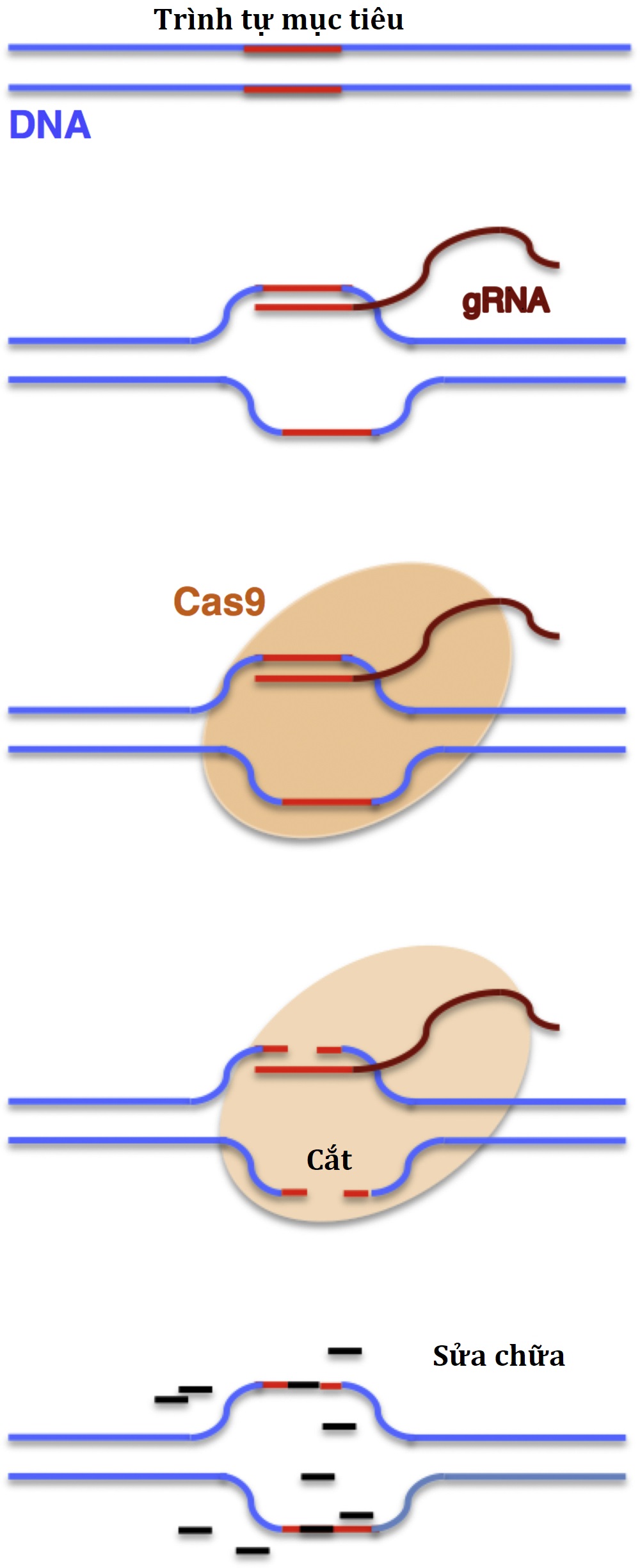
Hình 2.9 Hệ thống CRISPR-Cas9. Enzyme Cas9 hoạt động như một cặp ‘kéo phân tử’ có thể cắt hai sợi DNA tại một vị trí được chọn trước trong bộ gen, được dẫn đường tại chỗ bởi một trình tự RNA ngắn (gRNA). Cơ chế của tế bào sau đó sẽ hoàn thành quá trình sửa chữa DNA bằng cách đưa vào một đoạn tùy chọn hoặc phá vỡ trình tự mục tiêu.
Mặc dù hệ thống CRISPR-Cas9 có rất nhiều tiềm năng như một công cụ để chỉnh sửa bộ gen của các tế bào soma (không sinh sản), có nhiều tranh cãi liên quan đến tiềm năng chỉnh sửa các tế bào dòng mầm (sinh sản). Khả năng về mặt khái niệm bị hạn chế bởi các hàm ý đạo đức, vì những thay đổi gen sẽ được truyền lại cho các thế hệ tương lai của cá nhân được điều trị. Hơn nữa, có những lo ngại về tính đặc hiệu của gRNA trong việc dẫn đường một thay đổi có mục tiêu trong trình tự DNA quan tâm, vì một số dữ liệu chỉ ra rằng các đoạn DNA khác và/hoặc lớn hơn dự định có thể bị loại bỏ, đưa vào các thay đổi DNA không mong muốn (ngoài mục tiêu) có thể gây bệnh cho (các) tế bào mục tiêu. Ở một số quốc gia, ví dụ như Vương quốc Anh, việc thực hiện những thao tác trên dòng mầm này hiện tại bị cấm ở người. Tổ chức Y tế Thế giới đã triệu tập một hội đồng chuyên gia để thảo luận về vấn đề này và đưa ra các hướng dẫn thích hợp cho các cân nhắc lâm sàng có thể có trong tương lai.
DI TRUYỀN HỌC VỊ TRÍ TRONG NỘI TIẾT HỌC
Các nguyên tắc của Di truyền học Vị trí
Với mục đích xác định gen gây bệnh, phương pháp tiếp cận gen ứng cử viên dựa trên kiến thức một phần về cơ sở di truyền của bệnh đang được điều tra. Quá trình này đã thành công trong việc xác định các gen gây bệnh có chức năng rõ ràng. Ví dụ, các khiếm khuyết di truyền của hầu hết các rối loạn enzyme di truyền, bao gồm các hội chứng tăng sản tuyến thượng thận bẩm sinh (CAH), đã được biết đến vào cuối những năm 1980, khi việc giới thiệu PCR làm cho các công cụ của sinh học phân tử trở nên phổ biến trong cộng đồng nghiên cứu y học và di truyền. Tuy nhiên, vào khoảng cùng thời gian, nghiên cứu về các bệnh không có gen ứng cử viên rõ ràng (ví dụ, các hội chứng đa u tuyến nội tiết [MEN]) hoặc các bệnh mà việc sàng lọc các gen ứng cử viên rõ ràng không phát hiện được đột biến vẫn đang diễn ra. Chính trong những bệnh này, việc áp dụng “di truyền học ngược”, hay được gọi đúng hơn là dòng hóa vị trí (positional cloning), đã mang lại thông tin về cơ sở di truyền của các tình trạng bệnh lý này. Dòng hóa vị trí được bổ sung bởi Dự án Bộ gen Người (HGP) và mạng lưới toàn cầu trong việc cung cấp, một cách nhanh chóng và có kiểm soát, thông tin mà nếu không sẽ không thể tiếp cận được.
Quá trình của di truyền học vị trí (positional genetics) được trình bày trong Hình 2.10. Bước đầu tiên là thu thập thông tin lâm sàng từ các gia đình có thành viên bị ảnh hưởng, xác định phương thức di truyền của khiếm khuyết (trội hoặc lặn trên nhiễm sắc thể thường, liên kết X, di truyền phức tạp), và xác định kiểu hình của các đối tượng (hoặc mô), theo các tiêu chí đã được thiết lập rõ ràng cho việc chẩn đoán rối loạn. Nếu phương thức di truyền không được biết, cần phải thực hiện phân tích phân ly chính thức để xác định tính chất di truyền trên nhiễm sắc thể thường, hoặc liên kết X, và tính chất lặn hoặc trội. Một khi xác định này được thực hiện và độ xâm nhập (penetrance) của rối loạn được biết, phần mềm phân tích liên kết phù hợp có thể được sử dụng. Liên kết (Linkage) được kiểm tra bằng các dấu hiệu đa hình bao phủ toàn bộ bộ gen người; bất kỳ dấu hiệu nào cho thấy sự đa hình và được biết là nằm gần hoặc trong một gen gây bệnh giả định đều có thể được sử dụng. Liên kết di truyền có thể được định nghĩa là xu hướng các alen nằm gần nhau trên cùng một nhiễm sắc thể được truyền cùng nhau như một đơn vị nguyên vẹn qua quá trình giảm phân. Sức mạnh của liên kết sau đó có thể được sử dụng như một đơn vị đo lường để tìm ra khoảng cách di truyền giữa các locus khác nhau. Đơn vị khoảng cách bản đồ này là một ước tính gần đúng của khoảng cách vật lý nhưng cũng phụ thuộc nhiều vào các yếu tố khác (ví dụ, tần suất tái tổ hợp có sự khác biệt về giới tính, và khác nhau giữa các nhiễm sắc thể khác nhau và dọc theo chiều dài của một nhiễm sắc thể cụ thể). Phương pháp điểm số logarit của tỷ lệ odds (LOD) (logarithm of odds score) được sử dụng rộng rãi cho phân tích liên kết (linkage analysis).
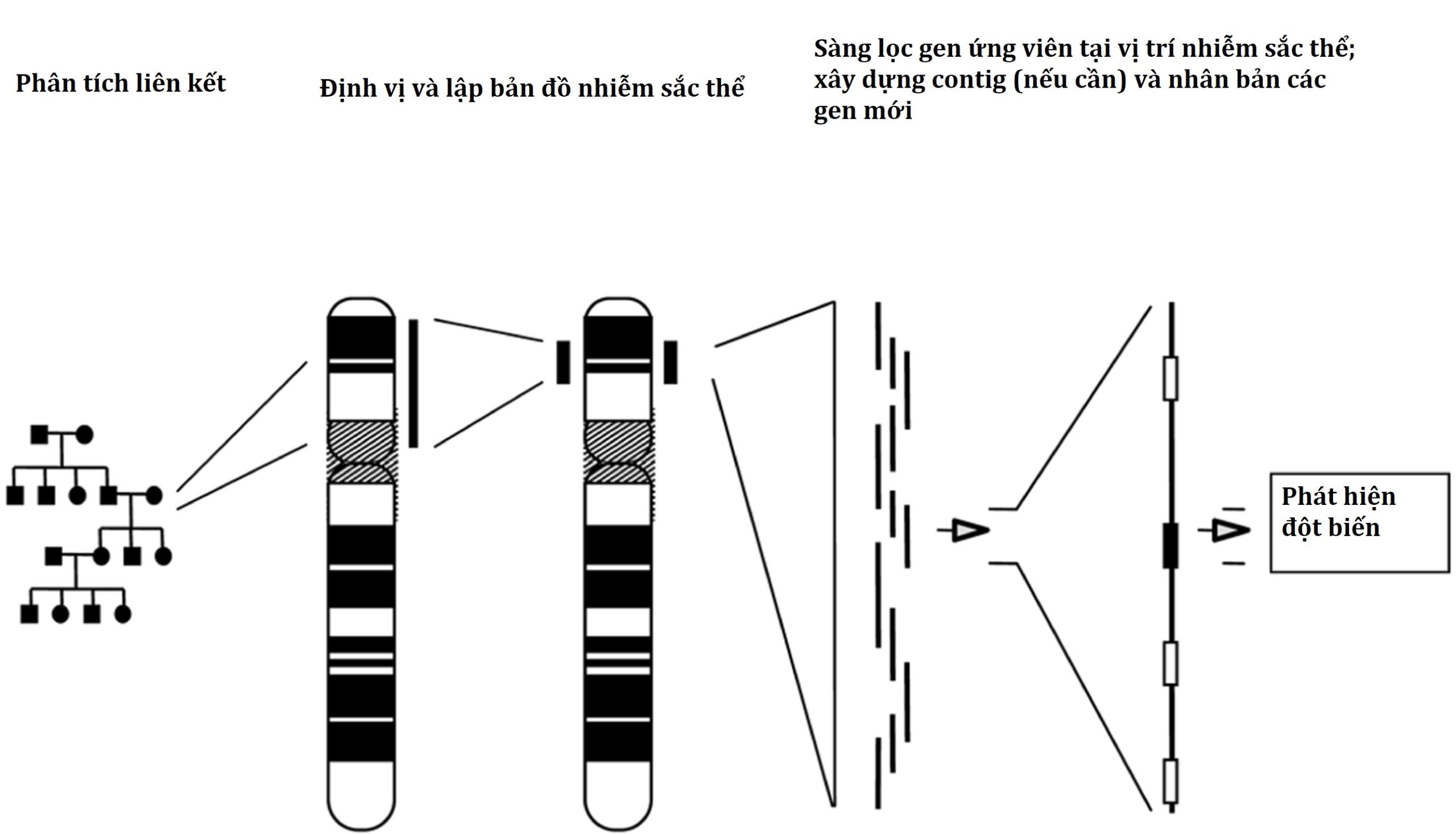
Hình 2.10 Các bước của dòng hóa vị trí.
Một khi một locus trên một nhiễm sắc thể đã được xác định, vùng này (thường dài vài nghìn cặp base) được thu hẹp bằng cách phân tích các tái tổ hợp có thông tin trong đoàn hệ bệnh nhân và gia đình có sẵn để nghiên cứu. Vùng bệnh có thể chứa các gen đã được lập bản đồ. Các cơ sở dữ liệu trực tuyến như GenBank, ENSEMBL (www.ensembl.org), và các cơ sở dữ liệu khác, và đặc biệt đối với các bác sĩ lâm sàng, là Di truyền Mendel ở Người Trực tuyến (OMIM), có thể cung cấp tất cả thông tin cần thiết. Nếu một bản sao là một ứng cử viên hợp lý, việc sàng lọc đột biến có thể xác định gen gây bệnh. Tuy nhiên, nếu những bước này không xác định được gen gây bệnh, có thể cần phải sàng lọc các trình tự mới từ khu vực đó; ngày nay, điều này thường được thực hiện bằng NGS, sau đó là giải trình tự Sanger để xác nhận. Các bản đồ nhiễm sắc thể được liên kết bởi các vị trí được đánh dấu bằng trình tự (STSs) có mặt trong nhiều hơn một bản sao bộ gen, do đó cung cấp thông tin quan trọng cho phép sắp xếp đúng DNA trong một locus nhất định. Các dấu hiệu đa hình (bao gồm cả những dấu hiệu được sử dụng cho phần phân tích liên kết của quá trình) là các STS hữu ích nhất vì chúng cung cấp một liên kết trực tiếp giữa dữ liệu lập bản đồ di truyền và vật lý. Các bản sao riêng lẻ có thể được giải trình tự; các gen được xác định trong quá trình này thông qua các đặc điểm trình tự độc đáo của chúng hoặc thông qua dịch mã in vitro. Trong quá khứ, các ESTs cung cấp thông tin về những trình tự gen nào được biểu hiện từ khu vực này. Ngày nay, gần như tất cả các gen đã được giải trình tự đầy đủ; tuy nhiên, các ESTs vẫn hữu ích để xem xét, đặc biệt là khi người ta đang cố gắng xác định một gen mới cho một locus liên quan đến bệnh nhất định. Mỗi một trong số các gen mới được xác định có thể được sàng lọc đột biến, miễn là hồ sơ biểu hiện của bản sao được xác định phù hợp với phổ của các mô bị ảnh hưởng bởi bệnh đang được điều tra. Mặc dù điều này hữu ích cho hầu hết các bệnh, đối với những bệnh khác, hồ sơ biểu hiện thậm chí có thể gây hiểu lầm; do đó, sự hiện diện của một bản sao trong một mô bị ảnh hưởng không phải lúc nào cũng cần thiết. Sự phân ly hoàn toàn của bệnh với một đột biến được xác định, bằng chứng chức năng, hoặc các đột biến ở hai hoặc nhiều gia đình mắc cùng một bệnh thường được yêu cầu làm bằng chứng hỗ trợ rằng trình tự được nhân bản là gen gây bệnh.
Nhận dạng Gen “Nội tiết” trên Toàn bộ Bộ gen
Vào những năm 1990, dòng hóa vị trí đã được sử dụng để xác định một số gen liên quan đến nội tiết học; ngày nay, điều này đang được thực hiện bằng NGS và các phương pháp toàn bộ bộ gen khác. Các hội chứng u nội tiết, mặc dù hiếm gặp và có tác động khiêm tốn đến thực hành lâm sàng nội tiết hàng ngày, là những ví dụ tinh túy về các bệnh có nguyên nhân phân tử được làm sáng tỏ bằng dòng hóa vị trí. Việc xác định các gen này đã được hỗ trợ rất nhiều bởi việc sử dụng mô khối u cho các nghiên cứu, chẳng hạn như mất dị hợp tử (LOH), lai so sánh bộ gen, và các ứng dụng lai tại chỗ phát huỳnh quang (FISH). Những kỹ thuật này đã thu hẹp các vùng nhiễm sắc thể được xác định về mặt di truyền và do đó tạo điều kiện thuận lợi cho việc xác định các gen chịu trách nhiệm; các nghiên cứu LOH rất quan trọng trong việc xác định các gen bệnh von Hippel-Lindau (VHL-elongin), MEN1 (menin), bệnh Cowden (PTEN), hội chứng Peutz-Jeghers (STK11/LKB1), và phức hợp Carney (PRKAR1A). NGS ngày nay được sử dụng để giải trình tự toàn bộ bộ gen (giải trình tự toàn bộ bộ gen [WGS] – whole-genome sequencing) hoặc chỉ các gen được biểu hiện (giải trình tự toàn bộ exome [WES] – whole exome sequencing) của một bệnh nhân khởi phát (hoặc các thành viên gia đình của anh ta) với mục đích xác định các đột biến gây ra một bệnh sau khi các phương pháp được mô tả ở đây đã xác định vị trí của một locus quan tâm. Trong trường hợp không có thông tin liên kết hoặc thông tin định vị khác (chẳng hạn, không có tiền sử gia đình, vật liệu khối u, hoặc các mẫu DNA bổ sung), DNA bộ gen của một cá nhân (hoặc mô có nguồn gốc từ cá nhân) có thể được giải trình tự bằng WGS hoặc WES. Tuy nhiên, điều này dẫn đến việc xác định nhiều đột biến và các biến thể khác cần phải được loại trừ để xác định (các) gen đặc hiệu cho bệnh. Một sơ đồ quy trình điển hình để phân tích các nghiên cứu như vậy được hiển thị trong Hình 2.11.
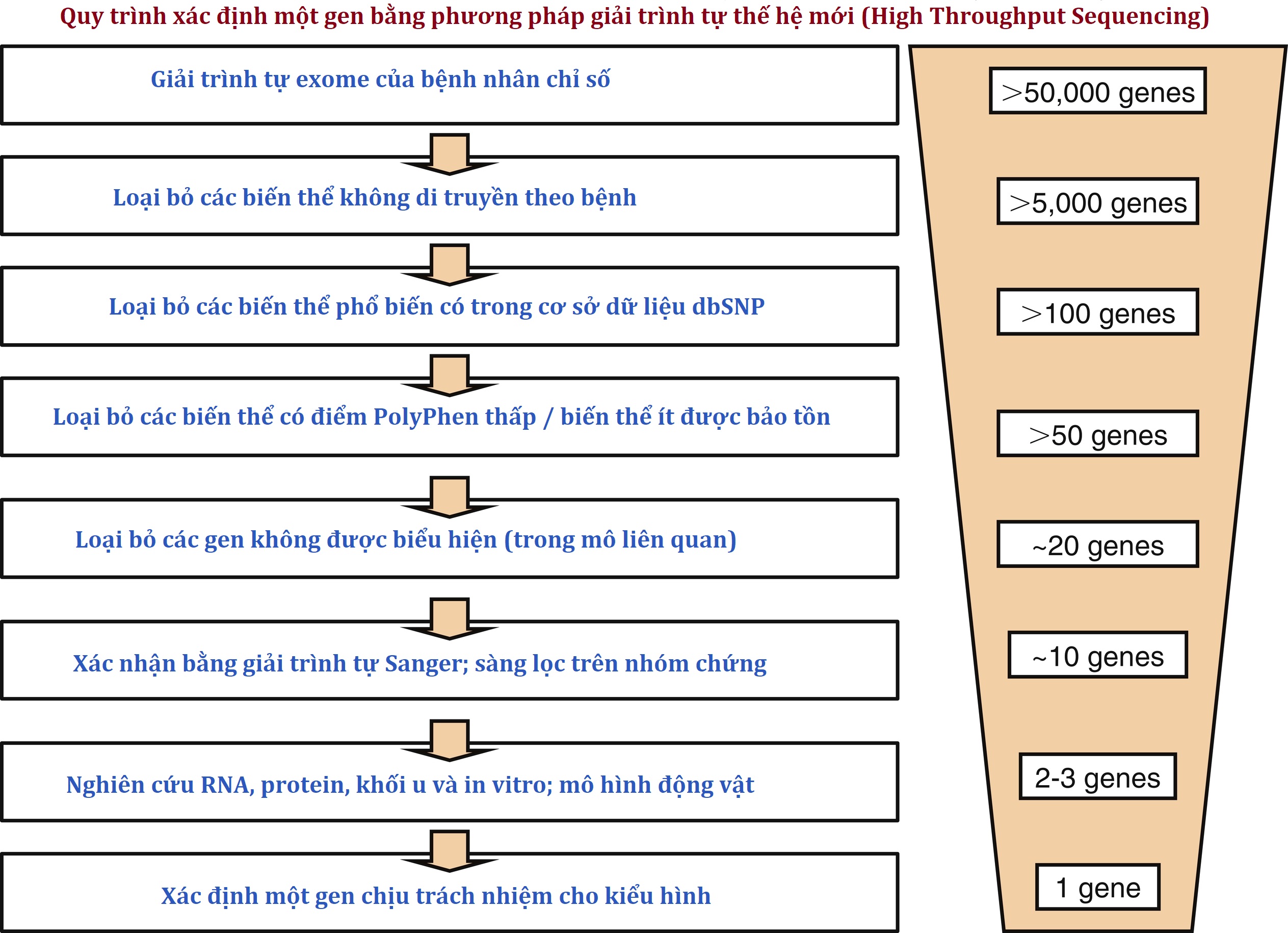
Hình 2.11 Giải trình tự toàn bộ bộ gen hoặc exome hiện là công nghệ giải trình tự DNA được ưa chuộng để xác định gen. Tuy nhiên, ngay cả trong bộ gen của những người có kiểu hình bình thường, các kỹ thuật giải trình tự này cũng cho ra một loạt các biến thể trình tự, bao gồm cả các đột biến cắt ngắn protein thực sự, do đó cần phải sử dụng một quy trình lọc có hệ thống và cẩn thận để xác định (các) gen gây bệnh. Các bước trong việc phân tích các đột biến gen được xác định bằng các kỹ thuật giải trình tự thông lượng cao, mà đỉnh điểm là việc gán một gen cho một kiểu hình cụ thể, được minh họa.
Tác động của Giải trình tự Hiện đại trong Thực hành Lâm sàng
Các nghiên cứu giải trình tự bộ gen chỉ ra rằng mỗi người mang tới 100 đột biến mất chức năng, với hơn 20 gen bị bất hoạt hoàn toàn. Nhiều biến thể di truyền được phát hiện có các chức năng dự phòng không ngờ tới trước đây; do đó một bệnh nhân có thể có nhiều biến thể di truyền trong một con đường tín hiệu duy nhất dẫn đến các kiểu hình được phân cấp.
Một ví dụ về việc áp dụng các kỹ thuật giải trình tự DNA hiện đại trong nội tiết nhi khoa dẫn đến việc làm sáng tỏ cơ chế bệnh là trong kiểu hình được gọi là bệnh Addison trắng (WAD) (white Addison disease). Người ta đã nhận ra rằng các bệnh nhân có biểu hiện suy thượng thận nguyên phát (pAI) không phải lúc nào cũng bị tăng sắc tố; biến thể này của pAI được gọi là WAD. Tăng sắc tố trong pAI được cho là do sự liên kết của nồng độ ACTH cao với thụ thể melanocortin (MC) 1 (MC1R). MC1R, một phân tử có độ tương đồng trình tự cao với MC2R, thụ thể ACTH, liên kết với phối tử thông thường của nó, hormone kích thích tế bào hắc tố α (α-MSH) và ACTH với ái lực gần như bằng nhau. α-MSH kích thích sản xuất melanin trong các tế bào hắc tố người được nuôi cấy và hoạt động đặc biệt để tăng tổng hợp eumelanin. Cả α-MSH và ACTH đều là các sản phẩm cắt nối của cùng một protein tiền thân ở tuyến yên, proopiomelanocortin (POMC), và đóng một vai trò trong việc xác định sắc tố ở người, vì các đột biến đồng hợp tử hoặc dị hợp tử phức hợp trong gen POMC có liên quan đến giảm sắc tố và tóc đỏ. Trong pAI không được điều trị thích hợp bằng glucocorticoid, biểu hiện POMC tăng lên, và cả α-MSH và ACTH đều tăng. Vai trò quan trọng của MC1R trong việc xác định sắc tố da và tóc của con người là không thể nghi ngờ: các alen MC1R có chức năng giảm dẫn đến tóc đỏ, tàn nhang, nhạy cảm với ánh nắng mặt trời, và tăng nguy cơ ung thư da, bao gồm cả u hắc tố.
Suy giảm glucocorticoid gia đình (FGD) (Familial glucocorticoid deficiency) do kháng ACTH bao gồm ít nhất ba hội chứng di truyền riêng biệt, tất cả đều di truyền theo kiểu lặn trên nhiễm sắc thể thường (AR): các đột biến bất hoạt của thụ thể ACTH (gen MC2R) và protein phụ của nó (MRAP); kháng ACTH đơn độc (IACTHR) không có đột biến MC2R, MRAP, hoặc bất kỳ đột biến nào khác đã biết; và hội chứng Allgrove. Turan và các đồng nghiệp đã mô tả một bệnh nhân mắc FGD không có tăng sắc tố: cô bé sinh ra với mái tóc đỏ, dần dần sẫm màu trong thời thơ ấu; mặc dù có các đợt hạ đường huyết lặp đi lặp lại, cô bé không được chẩn đoán mắc pAI và do đó đã tiếp xúc với nồng độ ACTH cao cho đến khi 6 tuổi. Giải trình tự MC2R cho thấy một đột biến đồng hợp tử T152K được biết là ảnh hưởng đến việc vận chuyển của thụ thể, giống như hầu hết các khiếm khuyết thụ thể ACTH gây ra FGD. Giải trình tự MC1R cho thấy một đột biến đồng hợp tử R160W là một trong những biến thể di truyền phổ biến nhất của thụ thể ở những người tóc đỏ. Trong một xác nhận hấp dẫn về vai trò của ACTH (và có thể cả các peptide khác có nguồn gốc từ POMC) và MC1R trong việc xác định không chỉ sắc tố da mà còn cả sắc tố tóc, tóc của bệnh nhân đã sáng lên và “trở lại màu hơi đỏ” sau khi được thay thế hợp lý bằng hydrocortisone và giảm nồng độ ACTH huyết thanh.
Với các biến thể di truyền chịu trách nhiệm cho các hiệu ứng thậm chí còn tinh vi hơn (so với, ví dụ, tóc đỏ), dữ liệu giải trình tự DNA thách thức bác sĩ lâm sàng thực hành phải tích hợp thông tin sinh học hệ thống vào thực hành lâm sàng. Rõ ràng không phải tất cả các khiếm khuyết trình tự được xác định bằng WGS hoặc WES đều gây ra bệnh; hoàn toàn ngược lại. Bằng chứng là có sự dư thừa và một sự cân bằng phân tử tinh tế và phức tạp đến lạ thường trong sinh học con người. Nhưng các bác sĩ lâm sàng phải kết hợp di truyền học vào thực hành hàng ngày của họ, và các nhà giáo dục phải đưa các con đường phân tử và sự biến đổi di truyền của chúng vào các bài giảng sinh lý học và sinh lý bệnh học kinh điển.
PHÂN TÍCH NHIỄM SẮC THỂ VÀ DI TRUYỀN HỌC TẾ BÀO PHÂN TỬ
Nhiễm sắc thể đại diện cho trạng thái cô đặc nhất trong quá trình biến thái của bộ gen trong một chu kỳ tế bào. Sự cô đặc của vật liệu di truyền ở giai đoạn kỳ giữa là một sự kiện quan trọng cung cấp sự phân ly chính xác và đồng đều của các nhiễm sắc thể giữa hai tế bào con mới sinh trong bước tiếp theo, kỳ sau. Điều này được theo sau bởi sự giãn ra của vật liệu di truyền sau khi tế bào phân chia. Khả năng biến đổi của bộ gen từ cấp độ phân tử (DNA) thành giai đoạn cận vi mô vật chất (nhiễm sắc thể) cung cấp một cơ hội duy nhất để hình dung bộ gen của một tế bào riêng lẻ của một sinh vật. Các bất thường nhiễm sắc thể khác nhau liên quan đến các bệnh hoặc hội chứng cụ thể có thể được phát hiện ở giai đoạn này bằng cách lập kiểu nhân đồ (karyotyping) (karyotyping) các nhiễm sắc thể.
Các nhiễm sắc thể có thể được nhận dạng và phân loại riêng lẻ theo kích thước, hình dạng (tỷ lệ cánh ngắn/dài), và bằng cách sử dụng các kỹ thuật nhuộm vi sai. Trong quá khứ, việc xác định các nhiễm sắc thể chỉ giới hạn ở các nhóm nhiễm sắc thể. Sự ra đời của kỹ thuật nhuộm băng nhiễm sắc thể đã cách mạng hóa phân tích di truyền tế bào. Các mẫu băng được đặt tên theo các chữ viết tắt sau: G cho Giemsa, R cho đảo ngược, Q cho quinacrine, và DAPI cho 4’6′-diamino-2-phenylindole; hai loại cuối cùng cho một mẫu tương tự như băng G. Sự phát triển hơn nữa của các kỹ thuật nhuộm băng có độ phân giải cao đã cho phép nghiên cứu các nhiễm sắc thể ở các giai đoạn sớm hơn của quá trình nguyên phân, kỳ đầu và kỳ trước giữa. Các nhiễm sắc thể dài hơn và có một mẫu băng phong phú hơn ở những giai đoạn đó, cung cấp chi tiết tuyệt vời cho việc xác định các sai lệch nhiễm sắc thể.
Sơ lược về các phương pháp
Chuẩn bị các nhiễm sắc thể chất lượng tốt là một nghệ thuật. Nhiều phương pháp khác nhau để phân lập nhiễm sắc thể đã được phát triển trong di truyền tế bào từ những năm 1960. Nguyên tắc chính đằng sau tất cả các phương pháp là hãm các tế bào ở kỳ giữa bằng cách phá vỡ thoi vô sắc của tế bào. Thoi vô sắc kỳ giữa là một cấu trúc bao gồm các sợi ống được hình thành trong tế bào mà các nhiễm sắc thể được gắn vào bởi các tâm động (centrosomes). Thoi vô sắc phân tách các nhiễm sắc thể thành hai tế bào con. Tác nhân thường được sử dụng để phá vỡ thoi vô sắc là Colcemid. Thời gian tiếp xúc với Colcemid thay đổi tùy thuộc vào hoạt động tăng sinh của tế bào. Các tế bào có chỉ số tăng sinh cao cần thời gian tiếp xúc ngắn hơn với nồng độ Colcemid cao, 0,1 đến 0,07 µg/mL trong 10 đến 20 phút. Các tế bào phát triển chậm đòi hỏi thời gian tiếp xúc lâu hơn, 1 đến 4 giờ hoặc qua đêm với nồng độ thấp hơn, 0,01 đến 0,05 µg/mL. Tiếp xúc kéo dài với Colcemid hoặc sử dụng nồng độ cao làm tăng tỷ lệ nhiễm sắc thể ở kỳ giữa muộn, dẫn đến việc rút ngắn các nhiễm sắc thể. Ngược lại, một thời gian tiếp xúc ngắn với nồng độ Colcemid cao làm giảm tổng sản lượng của các kỳ giữa. Tối ưu là đạt được sự cân bằng của các thông số này. Có một số sửa đổi bổ sung cho phép làm giàu các nhiễm sắc thể dài (kỳ trước giữa) bằng cách sử dụng các tác nhân ngăn chặn sự cô đặc của DNA, chẳng hạn như actinomycin D, ethidium bromide, hoặc BrDU. Các kỹ thuật đồng bộ hóa tế bào cũng có thể làm tăng đáng kể tổng sản lượng của các nhiễm sắc thể kỳ giữa.
Ứng dụng
Nhiễm sắc thể là vật liệu vô giá để đánh giá tính toàn vẹn của bộ gen và sự bảo tồn của nó ở cấp độ nhiễm sắc thể vi mô. Các lĩnh vực ứng dụng bao gồm chẩn đoán trước sinh, xét nghiệm di truyền của nhiều hội chứng gia đình, bao gồm ung thư, dòng hóa vị trí của các gen, và lập bản đồ vật lý (gán các gen trên các nhiễm sắc thể và các vùng dưới nhiễm sắc thể). Số lượng và hình thái của tất cả 23 cặp nhiễm sắc thể ở người có thể được kiểm tra bằng cách sử dụng nhuộm băng G vi sai của các nhiễm sắc thể thu được từ một mẫu máu ngoại vi. Các sai lệch về số lượng nhiễm sắc thể hoặc các thay đổi nhiễm sắc thể có thể nhìn thấy, chẳng hạn như chuyển vị, mất đoạn, và đảo đoạn liên quan đến các vùng mở rộng, có thể được phát hiện bằng phương pháp này. Những tiến bộ, chẳng hạn như lập kiểu nhân đồ quang phổ, cho phép hình dung tốt hơn về lệch bội và chuyển vị giữa các nhiễm sắc thể khác nhau. Các sắp xếp lại tinh vi, chẳng hạn như mất đoạn cận vi mô hoặc chuyển vị ẩn (sự trao đổi của các vùng telomere nhỏ ở đầu xa giữa hai nhiễm sắc thể không tương đồng), có thể được hình dung bằng cách sử dụng các mẫu dò cụ thể trong kỹ thuật lai tại chỗ phát huỳnh quang (FISH) (fluorescent in situ hybridization) (Hình 2.12).
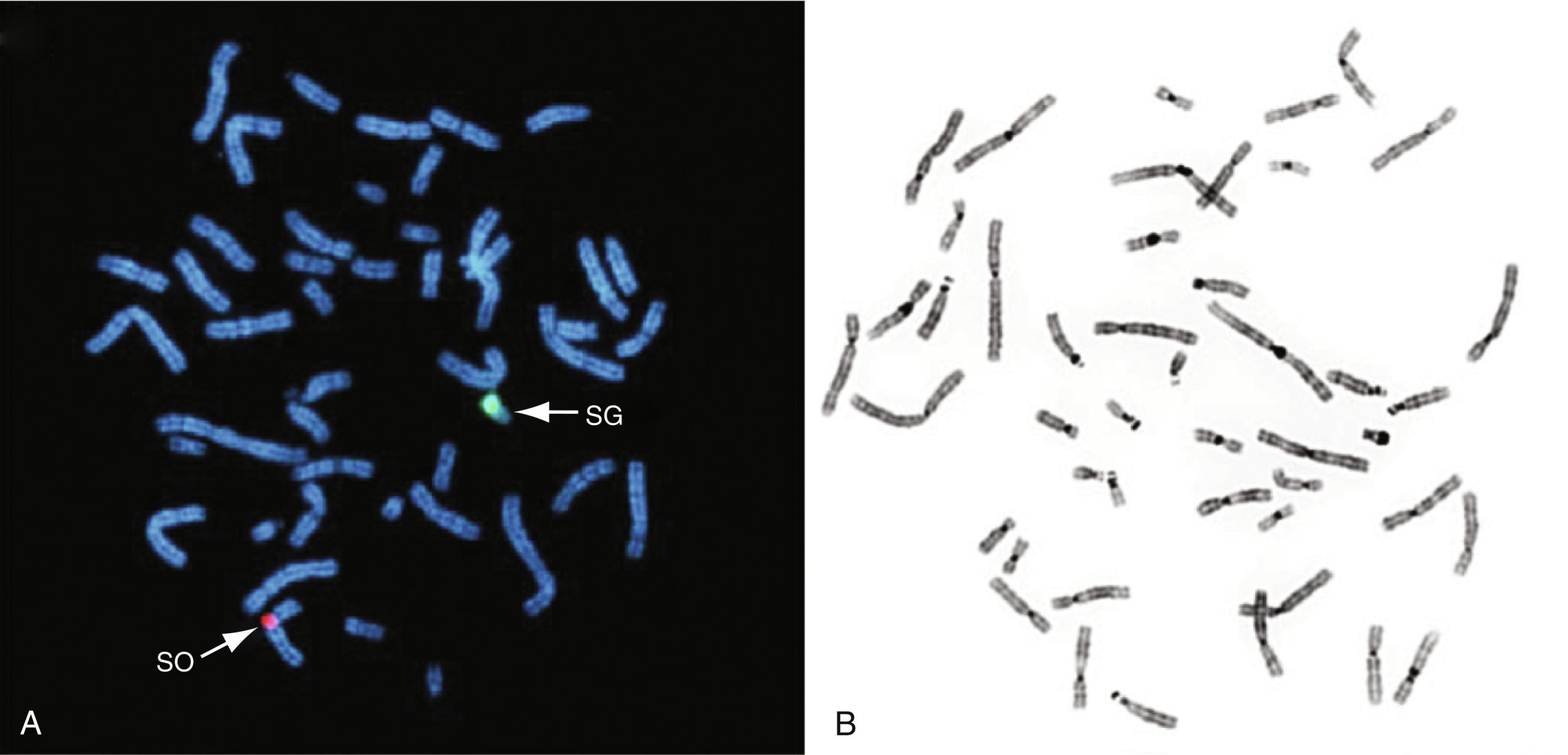
Hình 2.12 Nhiễm sắc thể kỳ giữa của người (A) sau khi lai tại chỗ phát huỳnh quang (FISH) sử dụng mẫu dò tâm động đặc hiệu cho nhiễm sắc thể X được đánh dấu bằng Spectrum Orange (SO) và dị nhiễm sắc đặc hiệu cho nhiễm sắc thể Y được đánh dấu bằng Spectrum Green (SG) và (B) với nhuộm băng DAPI đảo ngược (tương tự như băng G) cho phép nhận dạng nhiễm sắc thể.
Những phát triển trong tương lai
Phân tích nhiễm sắc thể sẽ vẫn là một công cụ phân tích mạnh mẽ trong các lĩnh vực lâm sàng và nghiên cứu trong tương lai gần. Các chiến lược khả thi để cải thiện các phương pháp hiện có bao gồm tự động hóa và tuyến tính hóa nội dung di truyền bằng cách tăng độ phân giải để hình dung ở cấp độ nhiễm sắc thể, chất nhiễm sắc, DNA, và gen. Một hướng phát triển khả thi khác là phân tích chức năng của bộ gen bằng cách sử dụng các nhiễm sắc thể cấu thành và các trình tự biểu hiện được đánh dấu từ các mô cụ thể được lập bản đồ trực tiếp đến vị trí ban đầu của chúng trên các nhiễm sắc thể.
Những phát triển trong tương lai
Phân tích nhiễm sắc thể sẽ vẫn là một công cụ phân tích mạnh mẽ trong các lĩnh vực lâm sàng và nghiên cứu trong tương lai gần. Các chiến lược khả thi để cải thiện các phương pháp hiện có bao gồm tự động hóa và tuyến tính hóa nội dung di truyền bằng cách tăng độ phân giải để hình dung ở cấp độ nhiễm sắc thể, chất nhiễm sắc, DNA, và gen. Một hướng phát triển khả thi khác là phân tích chức năng của bộ gen bằng cách sử dụng các nhiễm sắc thể cấu thành và các trình tự biểu hiện được đánh dấu từ các mô cụ thể được lập bản đồ trực tiếp đến vị trí ban đầu của chúng trên các nhiễm sắc thể.
NGUYÊN TẮC DIỄN GIẢI CÁC XÉT NGHIỆM DI TRUYỀN TRONG CHẨN ĐOÁN VÀ QUẢN LÝ CÁC BỆNH NỘI TIẾT NHI KHOA
Trong quá khứ, các xét nghiệm di truyền thường được thực hiện bởi các phòng thí nghiệm nghiên cứu có quan tâm cụ thể đến bệnh/hội chứng đang được điều tra. Trong kịch bản này, nhà cung cấp dịch vụ y tế nói chung có thể dựa vào chuyên môn của phòng thí nghiệm nghiên cứu để giúp xác định xét nghiệm/bảng xét nghiệm phù hợp và diễn giải kết quả của các xét nghiệm. Tuy nhiên, với việc ngày càng có nhiều phòng thí nghiệm thương mại thực hiện các xét nghiệm này, những trách nhiệm này đang được chuyển giao cho các nhà cung cấp dịch vụ y tế. Do đó, hiện nay các nhà cung cấp dịch vụ y tế cần phải làm quen với các vấn đề, chẳng hạn như lựa chọn xét nghiệm phù hợp, tính hữu ích tiềm tàng của thông tin do xét nghiệm cung cấp bao gồm kết quả dương tính giả và âm tính giả, các lựa chọn phòng ngừa hoặc điều trị có sẵn, và các vấn đề xã hội và hành vi liên quan đến xét nghiệm di truyền. Sau đây là một số điểm cần được xem xét khi yêu cầu hoặc diễn giải một xét nghiệm di truyền cho một rối loạn nội tiết nhi khoa:
Hạn chế của các xét nghiệm dựa trên PCR thường được sử dụng. Nói chung, các xét nghiệm dựa trên PCR thông thường (cho dù chúng liên quan đến điện di hay giải trình tự DNA của sản phẩm) không thể phân biệt một cách đáng tin cậy giữa hai alen của một gen riêng lẻ. Do đó, trong một trường hợp nhất định, việc phát hiện một đột biến trong một bệnh di truyền lặn trên nhiễm sắc thể thường có thể là do đột biến chỉ có ở một trong hai alen (và do đó không có khả năng có biểu hiện lâm sàng) hoặc là kết quả của việc đột biến có ở một alen duy nhất trong khi alen kia bị mất (ví dụ, do mất gen), trong trường hợp đó đột biến ở alen duy nhất có mặt sẽ biểu hiện lâm sàng. Tương tự, việc không thể khuếch đại một alen đột biến mất chức năng bằng PCR có thể là do sự hiện diện của hai alen bình thường (do đó loại trừ khiếm khuyết di truyền) hoặc do sự vắng mặt của cả hai alen (do mất gen), trong trường hợp đó khiếm khuyết di truyền sẽ có triệu chứng. Trong nhiều trường hợp, kịch bản sau có thể được loại trừ bằng cách xét nghiệm bằng các kỹ thuật thay thế, chẳng hạn như thấm Southern.
Đột biến dòng mầm so với đột biến soma. Đột biến dòng mầm (Germline mutations) có mặt trong mọi tế bào bắt nguồn từ hợp tử mà giao tử đột biến (trứng/tinh trùng) đó đã góp phần tạo nên. Ngược lại, đột biến soma (somatic mutations) xảy ra trong một tế bào soma (ví dụ, gan hoặc tủy xương hoặc da) và do đó không có mặt trong các loại tế bào khác trong cơ thể. Hầu hết các ví dụ phổ biến về đột biến di truyền là đột biến dòng mầm. Các ví dụ về đột biến soma gây ra các rối loạn nội tiết hiếm hơn—ví dụ, trong hội chứng McCune-Albright, đột biến trong gen GNAS có thể chỉ được phát hiện ở da (dát café-au-lait) hoặc xương (loạn sản xơ).
Rối loạn in dấu bộ gen. Trong nhiều trường hợp, cơ sở di truyền của rối loạn nội tiết không phải do một đột biến mà là do một bất thường trong in dấu bộ gen (genomic imprinting). In dấu bộ gen là sự điều chỉnh biểu hiện gen phụ thuộc vào việc vật liệu di truyền được thừa hưởng từ mẹ hay cha. Các ví dụ kinh điển là hội chứng Prader-Willi (mất đoạn nhiễm sắc thể 15q12 được thừa hưởng từ cha) và hội chứng Angelman (mất đoạn nhiễm sắc thể 15q12 được thừa hưởng từ mẹ). Các ví dụ khác về các rối loạn nội tiết có liên quan đến in dấu là giả suy cận giáp và loạn dưỡng xương di truyền Albright, hội chứng Russell-Silver, hội chứng Beckwith-Wiedemann, dạng khu trú của hạ đường huyết tăng insulin máu kéo dài ở trẻ sơ sinh, và đái tháo đường sơ sinh thoáng qua.
Độ xâm nhập và độ biểu hiện. Độ xâm nhập (Penetrance) được định nghĩa là tỷ lệ phần trăm những người có gen và những người phát triển kiểu hình tương ứng. Độ biểu hiện (Expressivity) là mức độ mà một gen được biểu hiện ở một người. Ví dụ, khi một gen có độ biểu hiện 50%, chỉ có một nửa các đặc điểm có mặt hoặc mức độ nghiêm trọng của bệnh chỉ bằng một nửa những gì có thể xảy ra với biểu hiện đầy đủ. Độ xâm nhập thay đổi của nhiều thành phần tân sinh của hội chứng MEN là một ví dụ về hiện tượng này.
Một gen, nhiều bệnh. Các ví dụ về các đột biến trong cùng một gen gây ra các bệnh khác nhau (ví dụ, đột biến gen lamin A/C [LMNA] gây ra loạn dưỡng cơ Emery-Dreifuss, lão nhi Hutchinson-Gilford, bệnh Charcot-Marie-Tooth loại 2, hội chứng loạn dưỡng mỡ cục bộ gia đình, và bệnh cơ tim giãn). Trong nhiều trường hợp như vậy, sự thay đổi có thể được cho là do các đột biến cụ thể cho mỗi biểu hiện lâm sàng khác nhau hoặc các bộ đột biến trong các miền/vùng khác nhau của gen. Một ví dụ tương tự là các đột biến trong gen tiền ung thư RET liên quan đến các hội chứng MEN2, u cận hạch không thuộc hội chứng, và trong bệnh Hirschsprung.
Một bệnh, nhiều gen (kiểu hình giả). Kiểu hình giả (Phenocopy) đề cập đến sự phát triển của các biểu hiện bệnh thường liên quan đến các đột biến của một gen cụ thể nhưng thay vào đó lại do một gen/nguyên nhân khác gây ra. Một kịch bản như vậy có thể làm rối loạn chẩn đoán lâm sàng và quản lý một rối loạn nội tiết di truyền bị nghi ngờ. Ví dụ, đã có báo cáo rằng hội chứng MEN1, thường do đột biến gen MEN1 gây ra, có thể bị bắt chước bởi tăng calci máu giảm calci niệu gia đình do đột biến bất hoạt trong thụ thể cảm nhận calci, hội chứng u hàm-cường cận giáp do đột biến gen chịu trách nhiệm cho cường cận giáp loại 2 (HRPT2), và các đột biến trong gen p27 (CDKN1B) gây ra một hội chứng giống MEN1 đã được gọi là MEN4.
Y HỌC BỘ GEN, GIẢI TRÌNH TỰ BỘ GEN, VÀ CÁC ỨNG DỤNG TRONG XÉT NGHIỆM DI TRUYỀN
Như đã đề cập trước đó, trong 3 thập kỷ qua, đã có hai tiến bộ lớn trong di truyền học phân tử hiện đại. Thứ nhất là về lý thuyết; đó là sự ra đời của khái niệm “dòng hóa vị trí”, đã được giải thích trước đó. Thứ hai là về kỹ thuật; kỹ thuật PCR cũng đã được thảo luận rộng rãi trong các trang trước. Y học ung thư và di truyền học người truyền thống là những lĩnh vực được hưởng lợi nhiều nhất từ các ứng dụng đầu tiên của các khái niệm và công nghệ bộ gen mới. Dự án Bộ gen Người (HGP) đã được hoàn thành vào năm 2003 bằng cách sử dụng chủ yếu giải trình tự Sanger dựa trên PCR. Phương pháp sau này đắt đỏ, tốn nhiều công sức và không thực tế để nghiên cứu toàn bộ bộ gen; do đó các công nghệ HGP phát triển từ sự cần thiết đã dẫn đến sự phát triển của các phương pháp NGS hiện đang được phổ biến rộng rãi. HGP cũng xác định rằng bộ gen người bao gồm 3,3 tỷ cặp base nucleotide và tất cả con người đều có 99,9% sự tương đồng ở cấp độ DNA với chỉ 0,1% biến thể di truyền, phần sau chủ yếu do đa hình nucleotide đơn (SNPs) (single-nucleotide polymorphisms) gây ra. Do đó có khoảng 10 triệu SNPs trong bộ gen người. Trung bình, các SNPs này xuất hiện một lần trong mỗi 300 nucleotide, chủ yếu trong DNA không mã hóa nằm giữa khoảng 20.000 đến 25.000 gen mã hóa. Nhiều thập kỷ sau những lần xác định gen gây bệnh thành công đầu tiên trong nội tiết học, các đột biến gen trong hội chứng CAH, các thụ thể insulin và hormone steroid, và RET, GNAS, menin, PTEN, và PRKARIA trong các dạng khác nhau của hội chứng u nội tiết đa tuyến, một số nguyên nhân di truyền khác đã được xác định trong các bệnh ảnh hưởng đến tuyến yên, tuyến giáp, tuyến cận giáp, tuyến tụy, tuyến thượng thận, tuyến sinh dục, v.v… Trên thực tế, tiến độ quá nhanh đến nỗi chúng ta đã nói về y học hoặc nội tiết học hậu bộ gen. Dù những khám phá này quan trọng đến đâu trong việc giúp chúng ta hiểu các quá trình tế bào, sự phát triển của tuyến, sinh lý bệnh, thậm chí trong một số trường hợp dẫn đến các phương pháp điều trị mới được thiết kế phân tử, chỉ đến bây giờ y học bộ gen mới thực sự làm thay đổi thực hành lâm sàng. Những thay đổi này nhanh chóng và sâu rộng, từ việc xác định kiểu gen của mỗi chúng ta, đến việc liên kết hồ sơ y tế điện tử (EMRs) với dữ liệu bộ gen, đến xét nghiệm di truyền trực tiếp đến người tiêu dùng (direct-to-consumer genetic testing – DCGT) và những tác động của nó đối với tương tác giữa bệnh nhân và bác sĩ, giám sát bệnh tật, đạo đức và hơn thế nữa. Hippocrates, một lần nữa, đã lưu ý rằng “Y học không thể được học nhanh chóng vì không thể tạo ra bất kỳ nguyên tắc đã được thiết lập nào trong đó, theo cách mà một người học viết theo một hệ thống mà mọi người dạy đều hiểu mọi thứ; vì tất cả những người hiểu chữ viết theo cùng một cách, làm như vậy bởi vì cùng một biểu tượng đôi khi không trở thành đối lập, mà luôn luôn kiên định giống nhau và không phụ thuộc vào sự tình cờ. Mặt khác, Y học không làm điều tương tự ở thời điểm này và thời điểm tiếp theo, và nó làm những điều đối lập với cùng một người, và vào lúc đó những điều tự mâu thuẫn.” Sự thay đổi liên tục của các ý tưởng và thực hành thực sự rất thực tế trong y học và nội tiết học hiện đại và được gây ra bởi những tiến bộ của di truyền học.
DI TRUYỀN HỌC TRONG THỰC HÀNH LÂM SÀNG VÀ CÁC ỨNG DỤNG GIẢI TRÌNH TỰ THẾ HỆ MỚI
Phần lớn bệnh nhân gặp trong thực hành nội tiết hàng ngày, cho đến gần đây, không phải là ứng cử viên cho xét nghiệm di truyền. Đối với hầu hết các bác sĩ nội tiết thực hành, việc gặp một bệnh nhân bị kháng glucocorticoid gia đình (FGR), ví dụ, gần như là chưa từng nghe thấy. Trong những trường hợp hiếm hoi này, việc chuyển đến một trung tâm học thuật nghiên cứu sẽ là giải pháp; ở đó, bệnh nhân của chúng ta, với FGR, sẽ được xét nghiệm di truyền có thể dẫn đến việc xác định đột biến gây bệnh trong gen thụ thể glucocorticoid. Khám phá này sẽ dẫn đến một bài báo nhưng sẽ vẫn tương đối mơ hồ đối với bác sĩ lâm sàng trung bình. Đây là trường hợp cho đến gần đây đối với hầu hết y học.
Tuy nhiên, ngày nay, có hơn 10.000 bệnh hiếm và mặc dù mỗi bệnh đều cực kỳ hiếm (như FGR), nhưng nói chung, những bệnh này ảnh hưởng đến số lượng lớn bệnh nhân và chiếm một tỷ lệ chi phí chăm sóc sức khỏe không cân xứng. Hiệp hội Nghiên cứu Bệnh hiếm Quốc tế ước tính rằng một số lượng đáng kể bệnh nhân mắc bệnh hiếm được khám và không được chẩn đoán tại các cơ sở lâm sàng. Một số bệnh nhân của chúng ta có các triệu chứng và dấu hiệu phức tạp hiện đang được đề nghị xét nghiệm di truyền trong các cơ sở lâm sàng thông thường. Ngoài ra, rất có thể hầu hết các bác sĩ lâm sàng ở các nước phát triển sẽ gặp những bệnh nhân đã nhận được DCGT. Vào tháng 2 năm 2019, ước tính có hơn 26 triệu người đã làm các xét nghiệm DNA tại nhà. Năm 2015, một công ty được biết đến với tên “23andme” đã được Cục Quản lý Thực phẩm và Dược phẩm Hoa Kỳ (FDA) cấp phép để bao gồm các đánh giá rủi ro sức khỏe trong các báo cáo di truyền. Ngày nay có một số công ty như vậy từ những công ty cung cấp DCGT về tổ tiên và các đặc điểm khác nhau, với một khoản phí tương đối rẻ, đến những công ty khác cung cấp dữ liệu phân tích trình tự bộ gen đầy đủ và thảo luận về các rủi ro sức khỏe với một mức giá cao hơn.
Trên một mặt trận khác, không có gì lạ khi một bác sĩ lâm sàng trung bình gặp phải dược lý di truyền (PGx) (pharmacogenomics). Ví dụ, khi xét nghiệm nồng độ cortisol bất thường để đáp ứng với nghiệm pháp ức chế dexamethasone 1 mg qua đêm (DEX), điều cần thiết là phải biết liệu bệnh nhân là người chuyển hóa DEX nhanh hay chậm. Hiện có một số nơi xét nghiệm các enzyme cytochrome P450 ở gan chịu trách nhiệm chuyển hóa DEX. Trong các lĩnh vực y học khác ngoài nội tiết học, PGx đã được áp dụng trong thực hành hàng ngày từ các bệnh truyền nhiễm và việc lựa chọn đúng kháng sinh cho người chuyển hóa nhanh hoặc chậm tương ứng, đến cách một số nhóm dân tộc phản ứng với một loại thuốc, dựa trên các biến thể di truyền đặc hiệu cho gen và liên quan đến dân tộc. Hiện có hơn 200 loại thuốc có thông tin PGx được bao gồm trong nhãn được FDA phê duyệt và các hướng dẫn thực hành lâm sàng mới, dựa trên PGx, hiện được xuất bản thường xuyên.
Cuối cùng, việc sử dụng sàng lọc di truyền để phòng ngừa sức khỏe và xét nghiệm chẩn đoán trước sinh ngày càng tăng. Có một số nỗ lực trên khắp Hoa Kỳ và các quốc gia khác và trong một số hệ thống y tế, tư nhân hoặc do chính phủ điều hành, để liên kết các yếu tố nguy cơ ung thư, tim mạch và các yếu tố nguy cơ khác với hồ sơ y tế (EMR và các loại hồ sơ khác) với mục đích dẫn đến các thay đổi về hành vi, chế độ ăn uống và các thay đổi khác, các thủ thuật chẩn đoán và thậm chí cả các thủ thuật xâm lấn nhằm phòng ngừa bệnh tật. Xét nghiệm tiền sản không xâm lấn (NIPT) (Noninvasive prenatal testing) dựa vào việc phát hiện DNA tự do ngoại bào (cfDNA) (cell-free DNA) trên các mẫu của cha mẹ để phát hiện các trisomy và ngày càng nhiều các khiếm khuyết di truyền khác. NIPT đại diện cho một trong những xét nghiệm được áp dụng nhanh nhất trong thực hành lâm sàng hàng ngày trong lịch sử y học và đã cách mạng hóa việc phát hiện các lệch bội của thai nhi và các rối loạn khác, loại bỏ nhu cầu chọc ối và một số thủ thuật khác.
Các chiến lược sàng lọc và xét nghiệm nói trên có thể thực hiện được là nhờ những tiến bộ bắt nguồn từ HGP, đặc biệt là các công nghệ giải trình tự DNA song song hàng loạt, được giới thiệu lần đầu tiên vào năm 2005 và không ngừng cải tiến và trở nên rẻ hơn mỗi ngày. Nói chung, các công nghệ giải trình tự DNA mới này có thể tạo ra hàng tỷ lần đọc trình tự ngắn trong vòng vài giờ; chi phí hiện tại cho việc xác định gần như toàn diện bộ gen của một người bằng NGS cho cả các biến thể thường xuyên và hiếm gặp là khoảng 1000 USD, trong khi chi phí thấp hơn đáng kể cho các phiên bản chỉ giải trình tự exome và thậm chí còn ít hơn nếu các vùng cụ thể hoặc một tập hợp các gen được nhắm mục tiêu.
Ngoài NGS, một sản phẩm của HGP là các vi mảng DNA mật độ cao dựa trên SNPs. Chúng được giới thiệu lần đầu tiên vào cuối những năm 1990; tuy nhiên, ban đầu, các vi mảng này chủ yếu được sử dụng trong các nghiên cứu dựa trên dân số hoặc các nghiên cứu đoàn hệ lớn khác về kiểu gen của bộ gen người cho các nghiên cứu về biến thể di truyền và các nghiên cứu liên kết khác. Bây giờ, các vi mảng DNA mật độ cao cũng được sử dụng trong thực hành lâm sàng để xác định các biến thể cấu trúc của bộ gen người và để xác định nhanh chóng bất kỳ biến thể trình tự nào: trên thực tế, các xét nghiệm di truyền phổ biến nhất hiện nay không phải là các xét nghiệm dựa trên giải trình tự, mà là các xét nghiệm lập kiểu gen sử dụng công nghệ có nguồn gốc từ các vi mảng DNA được các nhà nghiên cứu HGP phát triển lần đầu tiên. Thật khó để dự đoán NGS sẽ đi về đâu. Tuy nhiên, có thể nói một cách an toàn rằng nó sẽ mở rộng theo những cách và ứng dụng sẽ xâm nhập vào mọi khía cạnh của y học và thực sự là cuộc sống. Do đó, điều cần thiết đối với các bác sĩ nội tiết là phải hiểu những điều cơ bản về NGS, cách nó được áp dụng ngày nay, và tác động của nó đối với cách chúng ta thực hành y học.
Giải trình tự Bộ gen so với Exome, Diễn giải Biến thể, và Các Thách thức Khác
Mục tiêu của NGS là cung cấp một bản đồ chất lượng cao về sự biến đổi bộ gen cho mỗi mẫu. WGS cung cấp chính xác điều đó với công nghệ NGS đọc ngắn, hiện đã phổ biến. Tuy nhiên, nó vẫn còn quá đắt đỏ để sử dụng trong lâm sàng và cồng kềnh trong quản lý dữ liệu. Bởi vì gần 99% trong số khoảng 3,3 tỷ nucleotide tạo nên bộ gen người không mã hóa cho protein, và hầu hết các bệnh ở người là do các biến thể hoặc khiếm khuyết của các gen mã hóa (exome), phương pháp tiếp cận thay thế, như đã đề cập trước đó, là WES, hiện có giá dưới USD1000 và phổ biến ở các nước phát triển.
Các biến thể nucleotide đơn (SNVs) và các biến thể chèn hoặc mất đoạn nhỏ (indels) dài dưới 50 bp chiếm phần lớn các biến thể trong bộ gen người: có từ 3 đến 4 triệu SNVs và 0,4 đến 0,5 triệu indels trong mỗi mẫu người so với bộ gen tham chiếu. Tuy nhiên, chỉ có hơn 100 biến thể dẫn đến codon dừng sớm cho một gen mã hóa và chỉ khoảng 20 biến thể có khả năng gây hại. Tuy nhiên, thực tế mọi biến thể đều có thể gây ra một kiểu hình không chỉ thông qua việc thay đổi protein bằng cách thay thế một acid amin, mất đoạn, hoặc chèn đoạn, mà còn bằng cách thay đổi sự liên kết của các yếu tố phiên mã, tương tác với các phân tử khác, sự ổn định của RNA, v.v… Do đó, các biến thể trình tự được xác định bằng WGS hoặc WES được báo cáo là biến thể chưa rõ ý nghĩa (VUS) (variants of unknown significance) bởi vì trừ khi có dữ liệu tồn tại trước đó ghi nhận chức năng của chúng, chúng không thể được gọi là đột biến và không thể được liên kết chắc chắn với bệnh hoặc thậm chí là một yếu tố nguy cơ.
Biến thể cấu trúc (SV) (Structural variation) là một dạng biến đổi bộ gen khác, theo định nghĩa nó lớn hơn 50 bp, bao gồm biến thể số lượng bản sao (CNV) (copy number variants) đại diện cho sự khuếch đại hoặc mất đoạn của các đoạn DNA có kích thước thay đổi, các sắp xếp lại nhiễm sắc thể, và các chèn đoạn di động. SVs chỉ chiếm khoảng 0,2% tổng số biến thể, nhưng chúng thường xuyên ảnh hưởng đến kiểu hình hơn, ví dụ, chiếm từ 4% đến 12% các alen mã hóa có tác động cao. Hiệu ứng kiểu hình của chúng thường tỷ lệ thuận với kích thước của chúng, mặc dù có những ngoại lệ cho quy tắc này. SVs không dễ dàng phát hiện bằng WGS đọc ngắn và thậm chí còn khó hơn bằng WES. Sự đa dạng về kiến trúc của chúng cũng đặt ra những thách thức bổ sung trong việc phát hiện chúng. Để thấy được tác động của phương pháp luận, một bộ gen người điển hình chứa tới 10.000 SVs có thể phát hiện bằng WGS đọc ngắn thường được sử dụng nhưng hơn 20.000 SVs được xác định bằng WGS đọc dài. Sự phức tạp của việc diễn giải biến thể tăng lên khi người ta xem xét các yếu tố lặp lại, chẳng hạn như các đoạn lặp song song ngắn hoặc có số lượng thay đổi và các chèn đoạn di động.
Chú thích chức năng cho mỗi biến thể, cho dù đó là VUS, SV (CNV hay không), hoặc một biến thể lặp, được cập nhật liên tục dựa trên thông tin trong các cơ sở dữ liệu công khai mà dữ liệu kiểu gen và kiểu hình được thêm vào hàng ngày. Ước tính có tới 25% bệnh nhân mắc bệnh hiếm có thể được phát hiện có khiếm khuyết gây bệnh khi áp dụng và đọc kết quả xét nghiệm WES lần đầu tiên trong các đoàn hệ cụ thể được nghiên cứu bởi các trung tâm lâm sàng có kinh nghiệm. Do thông tin mới liên tục có sẵn, việc phân tích lại dữ liệu WES “âm tính” trước đây có thể làm tăng năng suất hơn 10% và thậm chí còn cao hơn khi dữ liệu gia đình và các dữ liệu khác được thêm vào phân tích.
Các biến thể có thể được phân loại là biến thể gây bệnh hoặc có khả năng gây bệnh và lành tính hoặc có khả năng lành tính dựa trên ảnh hưởng cấu trúc lên DNA và/hoặc gen hoặc protein mã hóa, các nghiên cứu chức năng, dữ liệu in silico, tần suất biến thể trong các quần thể đối chứng, các nghiên cứu gia đình (phân tích phân ly), và các yếu tố khác. Hiện có các hướng dẫn nghiêm ngặt và rõ ràng được tuân thủ bởi tất cả các phòng thí nghiệm xét nghiệm di truyền và được các hiệp hội chuyên môn tán thành. Việc đánh giá biến thể cũng bao gồm việc tìm kiếm cẩn thận trong y văn có sẵn nhưng người ta phải cẩn thận với danh pháp cũ, các ký hiệu gen khác nhau, cách đánh số trình tự, và các diễn giải kiểu hình.
Trong quá trình NGS, người ta cũng có thể xác định những gì đã được gọi là các phát hiện thứ cấp, điều này có thể hiểu được do số lượng các biến thể trong bộ gen người, như đã nêu trước đó. Hiện tại, Trường Cao đẳng Di truyền và Gen y học Hoa Kỳ (ACMGG) và các tổ chức khác đã xác định 59 gen có thể hành động về mặt y tế, trong đó, nếu phát hiện các biến thể, chúng cần được báo cáo là các phát hiện thứ cấp và có thể dẫn đến các xét nghiệm bổ sung và thậm chí cả các thủ thuật xâm lấn. Các ví dụ bao gồm các gen BRCA1 và BRCA2, liên quan đến ung thư vú, buồng trứng sớm và các bệnh ung thư khác; BMPRIA và SMAD4 liên quan đến tăng nguy cơ ung thư đại trực tràng và các bệnh ung thư khác; và các gen khác.
NGS và DNA tự do ngoại bào trong Y học Ung thư và Xét nghiệm Tiền sản
DNA trong huyết tương được mô tả lần đầu tiên vào năm 1948, nhưng trong nhiều năm nguồn gốc và ý nghĩa của nó không được biết đến. Vào những năm 1980, người ta nhận ra rằng các mô như khối u, nhau thai, và thai nhi người, thải ra DNA và RNA. Trong y học ung thư, cfDNA và cfRNA có thể dẫn đến phát hiện sớm, xác định hồ sơ đột biến của một khối u nhất định, và cung cấp phương tiện để theo dõi như một dấu ấn khối u và để đáp ứng với điều trị. Khái niệm “sinh thiết lỏng” (liquid biopsy) hiện đã được chấp nhận rộng rãi trong nhiều bối cảnh ung thư học mặc dù vẫn còn những thách thức: tỷ lệ cfDNA và cfRNA có nguồn gốc từ khối u tương đối thấp trong tuần hoàn, khó khăn trong việc đánh giá những bất thường như vậy sớm trong quá trình ác tính, và tỷ lệ dương tính giả cao.
Mặt khác, phân tích trình tự các đoạn cfDNA và cfRNA lưu thông trong máu của phụ nữ mang thai, cùng với việc chuyển đổi phương pháp này thành sàng lọc các bất thường nhiễm sắc thể của thai nhi, rõ ràng là một câu chuyện thành công lớn của di truyền học hiện đại: Kể từ cuối năm 2017, tổng cộng có từ 4 đến 6 triệu phụ nữ mang thai đã được phân tích DNA từ huyết tương của họ để sàng lọc lệch bội của thai nhi. Trong thai kỳ, các đoạn DNA được giải phóng từ nhau thai vào tuần hoàn của mẹ; huyết tương chứa cả DNA của mẹ và nhau thai và người ta tính toán tỷ lệ DNA của nhau thai so với tổng cfDNA, được gọi là “phân suất thai nhi” và tăng lên khi thai kỳ tiến triển. Việc xét nghiệm được thực hiện từ tuần thứ 10 của thai kỳ trở đi. Việc xét nghiệm đã cách mạng hóa việc phát hiện lệch bội và loại bỏ nhu cầu về các thủ thuật xâm lấn hơn (tức là, chọc ối). Tuy nhiên, có những vấn đề với tỷ lệ dương tính giả tương đối cao và việc xác định các phát hiện thứ cấp được điều chỉnh bởi cùng các hướng dẫn do ACMGG tài trợ đã được đề cập trong phần trước.
Giải trình tự thế hệ mới, thể khảm, và các ứng dụng trong việc phát hiện các khiếm khuyết di truyền soma
Một kết quả WGS hoặc WES “âm tính” trong một mẫu mô có thể do một số vấn đề đã được thảo luận trước đó và do vấn đề ngày càng thường xuyên của thể khảm (mosaicism): hầu hết các công nghệ NGS thương mại có sẵn có thể không phát hiện được thể khảm cho một biến thể gây bệnh vì biến thể sau có thể không có mặt trong mô được xét nghiệm (ví dụ, trong máu ngoại vi trong hầu hết các trường hợp). Việc chọn ra một biến thể ở mức độ thấp từ “nhiễu” của một trình tự thường là không thể (trừ khi người ta tăng “độ sâu” của trình tự), và mức độ thể khảm cho một biến thể thay đổi theo tuổi tác ở các mô khác nhau (các biến thể trình tự có thể phát hiện trong máu ở trẻ sơ sinh có thể không còn ở đó trong tuổi vị thành niên). Có những công cụ chẩn đoán mới đang được phát triển để phát hiện thể khảm, nhưng đối với hầu hết các trường hợp, nó vẫn là một vấn đề khó giải quyết.
Mặt khác, NGS được sử dụng thường xuyên để phát hiện phổ đột biến trong các bệnh ung thư. Các khối u có một bộ gen cực kỳ biến đổi, mang lại cơ hội chẩn đoán và điều trị nếu được xác định bằng NGS. Giống như những gì đã được đề cập trước đó trong phân tích cfDNA, các thách thức bao gồm tính không đồng nhất tương đối cao của các mẫu khối u, tần suất thấp của các thay đổi DNA đơn dòng, và tỷ lệ dương tính giả cao.
Trong các bệnh nội tiết, hội chứng McCune-Albright là một rối loạn kinh điển gây ra bởi thể khảm sau hợp tử cho một đột biến hoạt hóa của gen GNAS. NGS có thể được sử dụng để phát hiện thể khảm trong các mô ngoại vi, bởi vì thường thì đột biến không thể phát hiện được trong máu ngoại vi. Dự kiến rằng việc áp dụng rộng rãi hơn của NGS sẽ dẫn đến việc xác định nhiều trường hợp thể khảm hơn trong các bệnh nội tiết, giống như nó đã làm trong các lĩnh vực y học khác.
Y HỌC CHÍNH XÁC
Như đã lưu ý trong phần trước của chương này, xét nghiệm di truyền cho các rối loạn đơn gen, di truyền Mendel đang trở thành một công cụ ngày càng quan trọng đối với bác sĩ nội tiết nhi khoa. Nó có thể tự mình tiết lộ chẩn đoán và thông báo tiên lượng và điều trị. Ví dụ, một đột biến mất chức năng dị hợp tử trong ABCC8 được thừa hưởng từ người cha có thể là bằng chứng cho bệnh tăng insulin bẩm sinh do một tổn thương tụy khu trú có thể điều trị bằng cách cắt bỏ tổn thương, trong khi các đột biến mất chức năng dị hợp tử phức hợp trong cùng một gen được thừa hưởng từ cả cha và mẹ rất có thể sẽ cần phẫu thuật cắt bỏ gần toàn bộ tuyến tụy. Chẩn đoán đái tháo đường đơn gen có thể cho phép bệnh nhân trước đây được chẩn đoán mắc đái tháo đường type 1 chuyển từ insulin sang sulfonylurea đường uống. Ở các bé trai bị suy thượng thận nguyên phát, sự hiện diện của một đột biến gây bệnh trong ABCD1 là chẩn đoán cho bệnh loạn dưỡng chất trắng thượng thận và dự đoán một quỹ đạo tiên lượng và điều trị rất khác so với trường hợp do nguyên nhân tự miễn. Hơn nữa, các đột biến khác nhau trong cùng một gen có thể có những ý nghĩa khác nhau, như trong CAH gây ra bởi các đột biến trong CYP21A2, trong đó các kiểu hình mất muối, nam hóa, hoặc không cổ điển thường có thể được dự đoán bởi các đột biến cụ thể. Ngoài ra, khi các liệu pháp dựa trên gen và tế bào cụ thể cho các bệnh nội tiết xuất hiện, chẳng hạn như chỉnh sửa gen, chỉnh sửa base, và các liệu pháp oligonucleotide đối nghĩa (ASO) (anti-sense oligonucleotide), việc ghi nhận tổn thương di truyền chính xác của bệnh nhân sẽ trở nên quan trọng để thiết kế và cung cấp phương pháp điều trị thích hợp một cách chính xác. Các bác sĩ nội tiết nên hiểu cơ sở di truyền của các bệnh nội tiết và phương pháp điều trị của chúng để họ có thể tư vấn cho bệnh nhân, tham gia vào các quyết định điều trị, và theo dõi các hậu quả điều trị.
Tuy nhiên, mặc dù tầm quan trọng ngày càng tăng của xét nghiệm di truyền trong nội tiết nhi khoa, một số rào cản vẫn tồn tại. Đầu tiên và quan trọng nhất, nhiều bác sĩ còn thiếu kiến thức về y học di truyền. Hầu hết các bác sĩ, đặc biệt là những người đã tốt nghiệp trường y trong 25 năm qua, đã được giáo dục đầy đủ về di truyền cơ bản và lâm sàng, nhưng vì kiến thức này không được tiếp tục trong quá trình nội trú, đào tạo chuyên khoa, hoặc phát triển giảng viên, họ không cảm thấy thoải mái khi áp dụng các nguyên tắc di truyền lâm sàng vào thực hành lâm sàng. Các bác sĩ nên (1) thành thạo trong việc nhận ra khi nào một bệnh nhân có thể có cơ sở di truyền cho bệnh của họ khi nó xuất hiện ở độ tuổi rất trẻ, khi nó rất nặng, khi nó là một phần của một hội chứng, và khi sự di truyền của nó phân ly trong một gia đình; (2) nhận ra các kiểu di truyền khác nhau của trội trên nhiễm sắc thể thường, lặn trên nhiễm sắc thể thường, liên kết X, và di truyền in dấu từ mẹ hoặc cha; (3) nhận ra hậu quả của các đột biến mất chức năng, tăng chức năng, và trội âm, và rằng các đột biến khác nhau trong cùng một gen có thể có những hậu quả khác nhau; (4) nhận ra các bệnh nội tiết cụ thể có thể có cơ sở đơn gen, cũng như các gen chịu trách nhiệm; (5) hiểu các loại xét nghiệm di truyền có sẵn (gen đơn, bảng gen, exome, bộ gen, vi mảng, tế bào di truyền/kiểu nhân đồ, PCR, FISH, v.v…) và cách sử dụng chúng; (6) biết cách diễn giải các kết quả trình tự DNA khác nhau (lành tính so với gây bệnh so với ý nghĩa không chắc chắn); và (7) hiểu rằng việc diễn giải kết quả xét nghiệm của một bệnh nhân có thể thay đổi theo thời gian khi kiến thức mới xuất hiện, và biết cách liên hệ lại với phòng thí nghiệm xét nghiệm ban đầu và tìm kiếm các cơ sở dữ liệu công khai (ClinVar, Cơ sở dữ liệu Gen Đột biến Người, OMIM, v.v…) để biết thông tin mới.
Việc diễn giải đúng kết quả xét nghiệm di truyền bởi một phòng thí nghiệm xét nghiệm đòi hỏi nó phải có thông tin kiểu hình chính xác, phải được cung cấp bởi bác sĩ yêu cầu xét nghiệm. Đối với các xét nghiệm gen đơn và bảng gen, các loại đột biến khác nhau trong cùng một gen có những hậu quả khác nhau. Ví dụ, các biến thể khác nhau trong CASR gây ra hoặc là tăng calci máu giảm calci niệu gia đình (mất chức năng) hoặc hạ calci máu (tăng chức năng). Phòng thí nghiệm xét nghiệm sẽ miễn cưỡng diễn giải ý nghĩa của một biến thể mà không biết nên liên kết nó với kiểu hình nào. Khi đánh giá kết quả trình tự DNA exome hoặc bộ gen, phòng thí nghiệm xét nghiệm được hướng dẫn bởi kiểu hình (thường được ghi nhận bằng các thuật ngữ bản thể học kiểu hình người hoặc HPO) để chọn các biến thể trong số khoảng 20.000 gen người để đánh giá tính gây bệnh. Thông tin kiểu hình không chính xác hoặc không đầy đủ có thể làm sai lệch phân tích này.
Bệnh nhân (và nếu là trẻ em, người giám hộ của họ) nên cung cấp sự đồng ý có hiểu biết cho tất cả các xét nghiệm di truyền. Ngay cả đối với các xét nghiệm gen đơn và bảng gen, họ nên được thông báo về cách xét nghiệm có thể hỗ trợ chẩn đoán, tiên lượng, hoặc điều trị. Họ nên hiểu rằng một kết quả có thể không cung cấp thông tin, có thể cung cấp hoặc xác nhận một chẩn đoán, có thể có ý nghĩa không chắc chắn, hoặc có thể cần phân tích lại trong tương lai. Họ nên hiểu rằng nếu DNA của cha mẹ cũng được phân tích (ví dụ, một nghiên cứu bộ ba sử dụng DNA từ bệnh nhân và cả hai cha mẹ sinh học giả định), có khả năng mối quan hệ cha con không đúng có thể được tiết lộ. Họ nên hiểu rằng kết quả xét nghiệm di truyền có thể dẫn đến sự phân biệt đối xử trong việc mua bảo hiểm nhân thọ, hoặc cung cấp bằng chứng về bệnh đã có từ trước, mà trong một số kịch bản tương lai có thể dẫn đến việc từ chối bảo hiểm y tế. Bệnh nhân cũng nên nhận ra rằng các nghiên cứu exome và bộ gen có thể tiết lộ các phát hiện gây bệnh ngoài phạm vi mục đích của xét nghiệm, chẳng hạn như khuynh hướng ung thư, mà họ phải quyết định có được thông báo hay không. Cuối cùng, bệnh nhân nên hiểu trách nhiệm tài chính của họ đối với xét nghiệm di truyền, vì nó được các công ty bảo hiểm y tế chi trả một cách khác nhau. Trước khi một xét nghiệm di truyền được yêu cầu, nên tìm hiểu xem nhà cung cấp bảo hiểm y tế của bệnh nhân có chấp thuận xét nghiệm hay không, và nếu có, liệu bệnh nhân có phải chịu trách nhiệm về chi phí đồng chi trả hoặc khấu trừ hay không.
Khi bác sĩ thảo luận với bệnh nhân cả về khuyến nghị ban đầu để thực hiện xét nghiệm di truyền, cũng như kết quả xét nghiệm, thường rất hữu ích khi làm điều này cùng với một nhà tư vấn di truyền, người được đào tạo trong tất cả các lĩnh vực nói trên, bao gồm cả cách thảo luận hiệu quả các vấn đề này với bệnh nhân và gia đình của họ.
Các liệu pháp y học chính xác hứa hẹn rất lớn cho tương lai của nội tiết nhi khoa. Việc thay thế gen đột biến kết hợp với ghép tế bào gốc tự thân đang được nghiên cứu ở những bệnh nhân bị loạn dưỡng chất trắng thượng thận. Điều trị in vivo bệnh teo cơ tủy sống bằng phương pháp ASO gần đây đã được FDA chấp thuận. Trong tương lai, việc sửa chữa trực tiếp các khiếm khuyết gen của con người trong các tế bào soma in vivo có thể thực hiện được bằng các chiến lược chỉnh sửa gen và chỉnh sửa base có sự hỗ trợ của CRISPR. Kiến thức về cơ sở di truyền cụ thể của bệnh của bệnh nhân sẽ trở thành bước đầu tiên cần thiết trong việc xem xét các liệu pháp tiềm năng trong tương lai này.
CÔNG NGHỆ DNA TÁI TỔ HỢP VÀ ĐIỀU TRỊ CÁC BỆNH NỘI TIẾT NHI KHOA
Từ quan điểm điều trị, công nghệ DNA tái tổ hợp (recombinant DNA technology) có thể được khai thác để điều chỉnh liệu pháp dược lý theo kiểu gen của bệnh nhân (tức là, liệu pháp dược lý có mục tiêu), thao tác gen trong cơ thể người (liệu pháp gen), hoặc thiết kế các tế bào nhân sơ hoặc nhân thực để sản xuất các protein, chẳng hang như hormone, sau đó có thể được dùng để điều trị hoặc chẩn đoán. Trong khi liệu pháp dược lý có mục tiêu và liệu pháp gen chủ yếu bị giới hạn trong lĩnh vực nghiên cứu, việc sử dụng các hormone được sản xuất bằng công nghệ DNA tái tổ hợp đã được thiết lập vững chắc trong nội tiết học lâm sàng. Về mặt lịch sử, insulin là hormone đầu tiên được tổng hợp bằng công nghệ DNA tái tổ hợp được chấp thuận cho sử dụng lâm sàng. Hiện tại, một loạt các hormone tái tổ hợp bao gồm hormone tăng trưởng (GH), hormone tạo hoàng thể, hormone kích thích nang trứng, hormone kích thích tuyến giáp (TSH), hormone tuyến cận giáp, và erythropoietin đang được sử dụng lâm sàng hoặc đang trong giai đoạn thử nghiệm lâm sàng tiên tiến.
Trên cơ sở lý thuyết, có thể tổng hợp bất kỳ hormone protein nào có gen đã được nhân bản và trình tự DNA được xác định. Do đó, công nghệ DNA tái tổ hợp giúp có thể chèn gen mã hóa cho một hormone protein cụ thể vào một tế bào chủ sao cho protein được sản xuất bởi bộ máy tổng hợp protein của tế bào chủ. Protein được tổng hợp sau đó được tách ra khỏi phần còn lại của các protein tế bào chủ để thu được dạng tinh khiết của hormone quan tâm. Cả tế bào nhân sơ và nhân thực đều có thể đóng vai trò là tế bào chủ để sản xuất protein bằng công nghệ này. Bởi vì các sửa đổi sau dịch mã, chẳng hạn như glycosyl hóa, có thể cần thiết cho hoạt động tối ưu của một hormone protein, việc lựa chọn hệ thống tế bào cụ thể được sử dụng để sản xuất một hormone protein cụ thể là rất quan trọng. Các hệ thống tế bào nhân sơ, chẳng hạn như Escherichia coli, phù hợp để sản xuất các hormone protein không cần sửa đổi sau dịch mã, chẳng hạn như GH. Các hệ thống tế bào nhân thực, chẳng hạn như các tế bào buồng trứng chuột hamster Trung Quốc có khả năng sửa đổi protein sau dịch mã, rất hữu ích để sản xuất các hormone, chẳng hạn như TSH, đòi hỏi glycosyl hóa để có hiệu quả sinh học tối ưu. Ngoài ra, các tế bào nhân thực có khả năng tổng hợp các protein trải qua quá trình gấp nếp thích hợp, một bước không được thực hiện bởi các tế bào nhân sơ. Các ưu điểm của việc sử dụng DNA tái tổ hợp để sản xuất các protein này bao gồm khả năng cung cấp vô hạn một dạng protein có độ tinh khiết cao và không có nguy cơ nhiễm bẩn với các mầm bệnh sinh học liên quan đến việc chiết xuất protein từ mô người hoặc động vật. Ngoài ra, công nghệ này cho phép phát triển các chất tương tự và chất đối kháng hormone dễ dàng hơn nhiều so với các quy trình tổng hợp protein thông thường.
Ảnh hưởng của các yếu tố di truyền đối với sự chuyển hóa của các loại thuốc khác nhau là một hiện tượng đã được xác định rõ với tác động của các isoenzyme khác nhau của cytochrome p450 lên chu kỳ bán rã trong tuần hoàn của các loại thuốc, chẳng hạn như thuốc chống co giật, là một ví dụ kinh điển của sự tương tác này. Một ví dụ khác về vai trò của kiểu gen đối với việc lựa chọn liệu pháp dược lý là hiện tượng thiếu máu tan máu do thuốc ở bệnh nhân thiếu men glucose-6-phosphate dehydrogenase. Cuộc cách mạng bộ gen đã cho phép khai thác các phương pháp tiếp cận tính toán để xác định các đa hình của các gen đã biết mã hóa cho các protein có các đặc điểm chức năng khác nhau, như đã thảo luận trước đó. Các nhà điều tra hiện có thể dự đoán phản ứng của một cá nhân cụ thể đối với một nhóm thuốc/hóa chất. Cách tiếp cận dược lý bộ gen này đối với liệu pháp lâm sàng đã thành công trong việc chứng minh mối liên hệ giữa các đa hình cụ thể trong thụ thể β-adrenergic và phản ứng với các chất chủ vận β ở bệnh nhân hen phế quản, và các đa hình trong các thụ thể hydroxytryptamine và phản ứng với các thuốc an thần kinh. Việc áp dụng rộng rãi các công cụ của sinh học phân tử để làm sáng tỏ cơ sở phân tử của hoạt động của hormone cũng đã mang lại lợi ích bằng cách cho phép tùy chỉnh liệu pháp dược lý của các bệnh và hội chứng nội tiết dựa trên khiếm khuyết di truyền cá nhân cụ thể. Một ví dụ như vậy là báo cáo về liệu pháp dược lý có định hướng cho một trẻ sơ sinh có bộ phận sinh dục không rõ ràng, do đột biến trong thụ thể androgen. Ở trẻ sơ sinh này có đột biến M8071 trong thụ thể androgen, các nghiên cứu chức năng in vitro đã chỉ ra rằng thụ thể đột biến thể hiện mất khả năng liên kết với testosterone trong khi vẫn giữ được khả năng liên kết với dihydrotestosterone. Hơn nữa, sự liên kết khác biệt này cũng được phản ánh trong việc bảo tồn tốt hơn khả năng hoạt hóa chuyển đổi của dihydrotestosterone (DHT) so với testosterone. Những phát hiện in vitro này đã được khai thác để điều trị cho trẻ sơ sinh bằng DHT, dẫn đến việc phục hồi sự phát triển của bộ phận sinh dục nam. Trường hợp này minh họa rằng trong những trường hợp được chọn lọc, các xét nghiệm chức năng in vitro có thể giúp xác định các nhóm nhỏ bệnh nhân có bộ phận sinh dục không rõ ràng và không nhạy cảm với androgen sẽ đáp ứng với liệu pháp androgen có mục tiêu. Có thể dự đoán rằng trong những năm tới, nhiều ví dụ hơn về các chiến lược điều trị sáng tạo như vậy và các phác đồ điều trị hormone “tùy chỉnh” sẽ trở thành thông lệ và được thực hiện trong thực hành nội tiết nhi khoa lâm sàng.
KẾT LUẬN
Việc áp dụng công nghệ DNA tái tổ hợp đã dẫn đến sự gia tăng đáng kể trong sự hiểu biết của chúng ta về các quá trình sinh lý và các tình trạng bệnh lý. Những thành tựu, chẳng hạn như trình tự đầy đủ của bộ gen người ở những đối tượng bình thường, đã dẫn đến một sự thay đổi mô hình trong cách chúng ta suy nghĩ về các nguyên nhân di truyền và khuynh hướng mắc bệnh và trong việc phân tích chức năng của hormone và các protein liên quan. Trong mô hình truyền thống, các nhà điều tra tìm cách khám phá các gen mới hoặc phân tích chức năng của các protein đã biết cần phải dành một phần đáng kể thời gian của họ để tiến hành “nghiên cứu tại phòng thí nghiệm” trong các “phòng thí nghiệm ướt”. Cách tiếp cận mới trong kỷ nguyên “hậu bộ gen” này tận dụng sức mạnh chưa từng có của sinh học tính toán (computational biology) để “khai thác” các cơ sở dữ liệu về nucleotide, trình tự protein, và các cơ sở dữ liệu liên quan khác. Trong tương lai, hầu hết các nhà nghiên cứu sẽ làm việc với các mô hình trừu tượng và các bộ dữ liệu được lưu trữ trong các cơ sở dữ liệu máy tính. Do đó, những khám phá ban đầu về các gen mới hoặc các tương tác mới giữa các protein đã biết hoặc các con đường truyền tín hiệu nội bào có thể được thực hiện bằng cách sử dụng sức mạnh phân tích của các công cụ phần mềm tính toán (di truyền học chức năng – functional genomics); những hiểu biết ban đầu này sau đó có thể được xác minh và mở rộng bằng các phương pháp phòng thí nghiệm truyền thống. Các ưu điểm của mô hình mới này là rõ ràng với các phương pháp tiếp cận tính toán mất thời gian ngắn hơn đáng kể, với ít yêu cầu về nhân lực hơn, và có thể dễ dàng mở rộng phạm vi tìm kiếm để bao gồm nhiều phân tử và sinh vật (lập hồ sơ phát sinh loài). Một số cơ sở dữ liệu có thể truy cập qua web thuộc phạm vi công cộng hiện đang đóng vai trò là kho lưu trữ chính cho thông tin này. GenBank là kho lưu trữ chính cho thông tin trình tự và hiện đang được Viện Y tế Quốc gia hỗ trợ. Một trong những nguồn chính về vị trí vật lý, các đặc điểm lâm sàng, các kiểu di truyền, và các thông tin liên quan khác của các khiếm khuyết gen cụ thể là OMIM do Đại học Johns Hopkins ở Baltimore, Maryland điều hành. Đại học Johns Hopkins cũng vận hành Cơ sở dữ liệu Bộ gen trực tuyến, cho phép các nhà khoa học xác định các đa hình và xác định các liên hệ cho các mẫu dò gen và các công cụ nghiên cứu liên quan khác. Số lượng ngày càng tăng của các rối loạn nội tiết (và các rối loạn khác) có thể được cho là do những thay đổi trong trình tự nucleotide của các gen cụ thể cũng đã làm tăng sự cần thiết của việc có sẵn các xét nghiệm di truyền chính xác, đáng tin cậy và kịp thời, chẳng hạn như phát hiện đột biến. Một nguồn cho thông tin như vậy là một trang web hợp tác (www.genetests.org) duy trì một danh mục cập nhật các xét nghiệm thương mại và dựa trên nghiên cứu cho các rối loạn di truyền.
Với việc sử dụng rộng rãi các công cụ mạnh mẽ này trong các phòng thí nghiệm trên toàn thế giới, các gen đang được nhân bản và các bệnh di truyền đang được lập bản đồ với một tốc độ nhanh chóng. Trong tất cả những phát triển thú vị này, người ta vẫn cần phải ghi nhớ rằng trong khi khoa học “mới” này đã cho phép các lĩnh vực sinh học người trước đây không thể tiếp cận được thăm dò và nghiên cứu, vẫn còn rất nhiều điều cần được hiểu về các quá trình bệnh riêng lẻ. Do đó, hiện tại, chúng ta chỉ có một sự hiểu biết sơ bộ về mối tương quan giữa kiểu hình và kiểu gen trong nhiều bệnh di truyền phổ biến, chẳng hạn như CAH. Những khoảng trống này trong kiến thức của chúng ta đòi hỏi các bác sĩ lâm sàng phải thận trọng khi đưa ra các quyết định điều trị chỉ dựa trên cơ sở các nghiên cứu phân tử và di truyền. Điều này đặc biệt đúng trong lĩnh vực chẩn đoán trước sinh và khuyến nghị chấm dứt thai kỳ dựa trên phân tích di truyền. Khi chúng ta cải thiện sự hiểu biết của mình về cơ sở phân tử và di truyền của bệnh và chuyển đổi kiến thức này thành những lợi ích tại giường bệnh, chúng ta, cả với tư cách cá nhân và xã hội, phải nhận thức được các vấn đề quan trọng liên quan đến quyền riêng tư của dữ liệu sức khỏe và phải cảnh giác chống lại việc lạm dụng bằng cách tiết lộ không phù hợp kiến thức mạnh mẽ này.
TÀI LIỆU THAM KHẢO
- Bolander F.: Molecular Endocrinology . 2004 . Academic Press , 3rd ed
- Shupnik M.A.: Gene Engineering in Endocrinology . 2000 . New York: Garland Science ,
- Alberts B., et al.: Molecular Biology of the Cell , New York . Garland Science . 2000.
- Lodish H., et al.: Molecular Cell Biology . 5th ed. New York: W. H . Freeman . 2003.
- Roberts R.J.: Restriction enzymes and their isoschizomers . Nucleic Acids Res 1989; 17 (suppl): pp. 347-388.
- Nathans D., Smith H.O.: Restriction endonucleases in the analysis and restructuring of DNA molecules . Ann Rev Biochem 1975; 44: pp. 273-293.
- Southern E.M.: Detection of specific sequences among DNA fragments separated by gel electrophoresis . J Mol Biol 1975; 98: pp. 503-517.
- Waters D.L.E., Shapter F.M.: The polymerase chain reaction (PCR): general methods . Methods Mol Biol (Clifton, NJ) 2014; 1099: pp. 65-75.
- Ishmael F.T., Cristiana Stellato C.: Principles and applications of polymerase chain reaction: basic science for the practicing physician . Ann Allergy Asthma Immunol 2008; 101 (4): pp. 437-443.
- Kocova M., et al.: Detection of Y chromosome sequence in Turner’s syndrome by Southern blot analysis of amplified DNA . Lancet 1993; 342: pp. 140-143.
- Lehrach H.D., et al.: RNA molecular weight determinations by gel electrophoresis under denaturing conditions . Biochemistry 1977; 16: pp. 4743-4751.
- Thomas P.S.: Hybridization of denatured RNA and small DNA fragments transferred to nitrocellulose . Proc Natl Acad Sci USA. 1980; 77: pp. 5201-5205.
- Wagner E.M.: Monitoring gene expression: quantitative real-time Rt-PCR . Methods Mol Biol (Clifton, NJ) 2013; 1027: pp. 219-245.
- Freeman W.M., et al.: Quantitative RT-PCR: pitfalls and potential . BioTechniques 1999; 26 (1): pp. 112-122, 124–125.
- In Innis M.A., et al. (eds): PCR Protocols: A Guide to Methods and Applications . 1990. San Diego, CA: Academic Press ,
- Navarro E., et al.: Real-time PCR detection chemistry . Clin Chim Acta; Int J Clin Chem 2015; 439: pp. 231-250.
- Giulietti A., et al.: An overview of real-time quantitative PCR: applications to quantify cytokine gene expression . Methods (San Diego, Calif) 2001; 25 (4): pp. 386-401.
- Mohr A.M., Mott J.L.: Overview of MicroRNA biology . Semin Liver Dis 2015; 35 (1): pp. 3-11.
- Wójcicka A., et al.: Mechanisms in endocrinology: microRNA in diagnostics and therapy of thyroid cancer . Eur J Endocrinol 2016; 174 (3): pp. R89-R98.
- Reuter J.A., et al.: High-throughput sequencing technologies . Mol Cell 2015; 58 (4): pp. 586-597.
- Maxam A.M., Gilbert W.: Sequencing end-labeled DNA with base-specific chemical cleavages . Methods Enzymol 1980; 65: pp. 499-560.
- Sanger F., et al.: DNA sequencing with chain-terminating inhibitors . Proc Natl Acad Sci USA. 1977; 74: pp. 5463-5467.
- Marzancola M.G., et al.: DNA microarray-based diagnostics . Methods Mol Biol (Clifton, NJ) 2016; 1368: pp. 161-178.
- Harrington C.T., et al.: Fundamentals of pyrosequencing . Arch Pathol Lab Med 2013; 137 (9): pp. 1296-1303.
- Choi M., et al.: K + Channel mutations in adrenal aldosterone-producing adenomas and hereditary hypertension . Science (New York, NY) 2011; 331 (6018): pp. 768-772.
- Zhang F., et al.: CRISPR/Cas9 for genome editing: progress, implications and challenges . Hum Mol Genet 2014; 23 (R1): pp. R40-R46.
- Collins F.S.: Positional cloning: let’s not call it reverse anymore . Nat. Genet 1992; 1: pp. 3-6.
- Collins F.S.: Positional cloning moves from perditional to traditional . Nat Genet 1995; 9: pp. 347-350.
- Borsani G., et al.: A practical guide to orient yourself in the labyrinth of genome databases . Hum Mol Genet 1998; 7: pp. 1641-1648.
- Khoury M.J., et al.: Fundamentals of Genetic Epidemiology , 1st ed. Oxford, UK . Oxford University Press . 1993.
- Speer M.C.: Use of LINKAGE programs for linkage analysis . Curr Protocol Hum Genet 2006; 1(1.7) .
- “Online Mendelian Inheritance in Man, OMIM.” National Library of Medicine, McKusick-Nathans Institute for Genetic Medicine, Johns Hopkins University (Baltimore, MD) and National Center for Biotechnology Information, National Library of Medicine (Bethesda, MD) : Center for Medical Genetics . Johns Hopkins University and National Ce . 2007. http://www.ncbi.nlm.nih.gov/omim/ .
- Linehan W.M., et al.: Identification of the von Hippel-Lindau (VHL) gene . JAMA 1995; 273: pp. 564-570.
- Chandrasekharappa S.C., et al.: Positional cloning of the gene for multiple endocrine neoplasia-Type 1 . Science 1997; 276: pp. 404-407.
- Liaw D., et al.: Germline mutations of the PTEN gene in Cowden disease, an inherited breast and thyroid cancer syndrome . Nat Genet 1997; 16: pp. 64-67.
- Hemminki A., et al.: A serine/threonine kinase gene defective in Peutz-Jeghers syndrome . Nature 1998; 391: pp. 184-187.
- Jenne D.E., et al.: Peutz-Jeghers syndrome is caused by mutations in a novel serine threonine kinase . Nat Genet 1998; 18: pp. 38-43.
- Kirschner L.S., et al.: Mutations of the gene encoding the protein kinase A type i-alpha regulatory subunit in patients with the Carney complex . Nat Genet 2000; 26: pp. 89-92.
- Genomes Project Consortium, Adam Auton, et al : A global reference for human genetic variation . Nature 2015; 526 (7571): pp. 68-74.
- Genomes Project Consortium, Gonçalo, R. A., et al : A map of human genome variation from population-scale sequencing . Nature 2010; 467 (7319): pp. 1061-1107.
- Turan S., et al.: An atypical case of familial glucocorticoid deficiency without pigmentation caused by coexistent homozygous mutations in MC2R (T152K) and MC1R (R160W) . J Clin Endocrinol Metabol 2012; 97 (5): pp. E771-E774.
- Millington G.W.M.: Proopiomelanocortin (POMC): the cutaneous roles of its melanocortin products and receptors . Clin Exp Dermatol 2006; 31 (3): pp. 407-412.
- Rees J.L., Harding R.M.: Understanding the evolution of human pigmentation: recent contributions from population genetics . J Invest Dermatol 2012; 132 (3 Pt 2): pp. 846-853.
- Clark A.J.L., et al.: The genetics of familial glucocorticoid deficiency . Best Pract Res Clin Endocrinol Metabol 2009; 23 (2): pp. 159-165.
- Brooks B.P., et al.: Genotypic heterogeneity and clinical phenotype in triple a syndrome: a review of the NIH Experience 2000-2005 . Clin Genet 2005; 68 (3): pp. 215-221.
- Stratakis C.A.: ‘Patients can have as many gene variants as they damn well please’: why contemporary genetics presents us daily with a version of Hickam’s Dictum . J Clin Endocrinol Metabol 2012; 97 (5): pp. E802-E804.
- Caspersson T., et al.: Analysis of human metaphase chromosome set by aid of DNA-binding fluorescent agents . Exp Cell Res 1999; 253 (2): pp. 302-304.
- Yunis J.J., Lewandowski R.C.: High-resolution cytogenetics . Birth Defects Orig Art Ser 1983; 19 (5): pp. 11-37.
- Yunis J.J.: High resolution of human chromosomes . Science 1976; 191: pp. 1268-1270.
- Mateuca R.A., et al.: Cytogenetic methods in human biomonitoring: principles and uses . Methods Mol Biol (Clifton NJ) 2012; 817: pp. 305-334.
- Green E.D., et al.: The future of DNA sequencing . Nature 2017; 550 (7675): pp. 179-181.
- Stratakis C.A.: Clinical genetics of multiple endocrine neoplasias, Carney Complex and related syndromes . J Endocrinol Invest 2001; 24 (5): pp. 370-383.
- Stratakis C.A.: Genetics and the New (Precision) Medicine and Endocrinology: in Medias Res or Ab Initio? . Endocrinol Metabol Clin North Am 2017; 46 (2): pp. xv-xvi.
- Potter P.: Hippocrates “Places in Man.” . 1995 . Cambridge, MA: Harvard University Press ,
- Stratakis, C.A., et al. (1994). Glucocorticosteroid resistance in humans. elucidation of the molecular mechanisms and implications for pathophysiology. Ann N. Y. Acad Sci , 746, 362–374; discussion 374–376.
- Boycott K.M., et al.: A diagnosis for all rare genetic diseases: the horizon and the next frontiers . Cell 2019; 177 (1): pp. 32-37.
- Crow D.: A new wave of genomics for all . Cell 2019; 177 (1): pp. 5-7.
- Abul-Husn N.S., Kenny E.E.: Personalized medicine and the power of electronic health records . Cell 2019; 177 (1): pp. 58-69.
- Sirugo G., et al.: The missing diversity in human genetic studies . Cell 2019; 177 (4): pp. 1080.
- Bianchi D.W., Chiu R.W.K.: Sequencing of circulating cell-free DNA during pregnancy . N Engl J Med 2018; 379 (5): pp. 464-473.
- Shendure J., et al.: Genomic medicine-progress, pitfalls, and promise . Cell 2019; 177 (1): pp. 45-57.
- Lappalainen T., et al.: Genomic analysis in the age of human genome sequencing . Cell 2019; 177 (1): pp. 70-84.
- Richards S., et al.: Standards and guidelines for the interpretation of sequence variants: a joint consensus recommendation of the American College of Medical Genetics and Genomics and the Association for Molecular Pathology . Genet Med 2015; 17 (5): pp. 405-424.
- Salpea P., Stratakis C.A.: Carney complex and McCune Albright syndrome: an overview of clinical manifestations and human molecular genetics . Mol Cell Endocrinol 2014; 386 (1–2): pp. 85-91.
- Eichler F., et al.: Hematopoietic stem-cell gene therapy for cerebral adrenoleukodystrophy . N Engl J Med. 2017; 377 (17): pp. 1630-1638.
- Chiriboga C.A., et al.: Results from a phase 1 study of nusinersen (ISIS-SMN(Rx)) in children with spinal muscular atrophy . Neurology 2016; 86 (10): pp. 890-897.
- Sheridan C.: Roche’s splashes USD2.4. billion on foundation medicine’s cancer platform . Nat Biotechnol 2018; 36 (8): pp. 669-670.
- Rees H.A., Liu D.R.: Base editing: precision chemistry on the genome and transcriptome of living cells . Nat Rev Genet 2018; 19 (12): pp. 770-788.
- Goeddel D.V., Kleid D.G., et al.: Expression in Escherichia Coli of chemically synthesized genes for human insulin . Proc Natl Acad Sci USA 1979; 76 (1): pp. 106-110.
- Riggs A.D.: Bacterial production of human insulin . Diabetes Care 1981; 4 (1): pp. 64-68.
- Goeddel D.V., Heyneker H.L., et al.: Direct expression in Escherichia Coli of a DNA sequence coding for human growth hormone . Nature 1979; 281 (5732): pp. 544-548.
- Gesundheit N., Weintraub B.D.: Mechanisms and regulation of TSH glycosylation . Adv Exp Med Biol 1986; 205: pp. 87-105.
- Ong Y.C., et al.: Directed pharmacological therapy of ambiguous genitalia due to an androgen receptor gene mutation . Lancet 1999; 354: pp. 1444-1445.
- MacArthur D.G., et al.: A systematic survey of loss-of-function variants in human protein-coding genes . Science (New York, NY) 2012; 335 (6070): pp. 823-828.
- Hsu S.Y., Hsueh A.J.W.: Discovering new hormones, receptors, and signaling mediators in the genomic era . Mol Endocrinol 2000; 14 (5): pp. 594-604.
BẢNG CHÚ GIẢI THUẬT NGỮ Y HỌC ANH VIỆT – CHƯƠNG 2
| STT | Thuật ngữ tiếng Anh | Phiên âm IPA | Nghĩa Tiếng Việt |
|---|---|---|---|
| 1 | Molecular endocrinology | /məˈlɛkjʊlər ˌɛndoʊkrɪˈnɒlədʒi/ | Nội tiết học phân tử |
| 2 | Endocrine genetics | /ˈɛndoʊkrɪn dʒəˈnɛtɪks/ | Di truyền học nội tiết |
| 3 | Genetic counseling | /dʒəˈnɛtɪk ˈkaʊnsəlɪŋ/ | Tư vấn di truyền |
| 4 | Inherited endocrine disorders | /ɪnˈhɛrɪtɪd ˈɛndoʊkrɪn dɪsˈɔːrdərz/ | Các rối loạn nội tiết di truyền |
| 5 | Endocrine oncology | /ˈɛndoʊkrɪn ɒŋˈkɒlədʒi/ | Ung thư học nội tiết |
| 6 | Thyroid cancer | /ˈθaɪrɔɪd ˈkænsər/ | Ung thư tuyến giáp |
| 7 | Monoclonal antibody | /ˌmɒnoʊˈkloʊnəl ˈæntɪˌbɒdi/ | Kháng thể đơn dòng |
| 8 | Fibroblast growth factor-23 (FGF-23) | /ˈfaɪbroʊˌblæst ɡroʊθ ˈfæktər twɛnti θriː/ | Yếu tố tăng trưởng nguyên bào sợi-23 |
| 9 | X-linked hypophosphatemic rickets | /ɛks-lɪŋkt ˌhaɪpoʊˌfɒsfəˈtiːmɪk ˈrɪkɪts/ | Còi xương hạ phosphat máu liên kết X |
| 10 | Molecular biology | /məˈlɛkjʊlər baɪˈɒlədʒi/ | Sinh học phân tử |
| 11 | Pharmacogenetics | /ˌfɑːrməkoʊdʒəˈnɛtɪks/ | Dược lý di truyền |
| 12 | Deoxyribonucleic acid (DNA) | /diˌɒksiˌraɪboʊnjuˌkleɪɪk ˈæsɪd/ | Acid deoxyribonucleic (DNA) |
| 13 | Nuclear proteins | /ˈnjuːkliər ˈproʊtiːnz/ | Protein nhân |
| 14 | White blood cells | /waɪt blʌd sɛlz/ | Tế bào bạch cầu |
| 15 | Anticoagulation | /ˌæntiˌkoʊˌæɡjʊˈleɪʃən/ | Chống đông máu |
| 16 | Lymphocytes | /ˈlɪmfoʊˌsaɪts/ | Tế bào lympho |
| 17 | Epstein-Barr virus | /ˈɛpstaɪn bɑːr ˈvaɪrəs/ | Virus Epstein-Barr |
| 18 | Cell culture | /sɛl ˈkʌltʃər/ | Nuôi cấy tế bào |
| 19 | Immortal cell lines | /ɪˈmɔːrtl sɛl laɪnz/ | Dòng tế bào bất tử |
| 20 | Molecular genetic studies | /məˈlɛkjʊlər dʒəˈnɛtɪk ˈstʌdiz/ | Nghiên cứu di truyền phân tử |
| 21 | Fibroblast-derived cultures | /ˈfaɪbroʊˌblæst dɪˈraɪvd ˈkʌltʃərz/ | Dòng nuôi cấy từ nguyên bào sợi |
| 22 | Ribonucleic acid (RNA) | /ˌraɪboʊnjuˌkleɪɪk ˈæsɪd/ | Acid ribonucleic (RNA) |
| 23 | Gene expression | /dʒiːn ɪkˈsprɛʃən/ | Biểu hiện gen |
| 24 | Messenger RNA (mRNA) | /ˈmɛsɪndʒər ɑːr ɛn eɪ/ | RNA thông tin (mRNA) |
| 25 | Polymerase chain reaction (PCR) | /pəˈlɪməreɪz tʃeɪn riˈækʃən/ | Phản ứng chuỗi polymerase (PCR) |
| 26 | Autosomal chromosome | /ˌɔːtəˈsoʊməl ˈkroʊməˌsoʊm/ | Nhiễm sắc thể thường |
| 27 | Haploid human genome | /ˈhæplɔɪd ˈhjuːmən ˈdʒiːnoʊm/ | Bộ gen đơn bội của người |
| 28 | Base pairs | /beɪs pɛərz/ | Cặp base |
| 29 | Restriction endonucleases | /rɪˈstrɪkʃən ˌɛndoʊˈnjuːklieɪzɪz/ | Endonuclease giới hạn |
| 30 | Recognition sites | /ˌrɛkəɡˈnɪʃən saɪts/ | Vị trí nhận biết |
| 31 | Bacteriophage | /bækˈtɪərioʊˌfeɪdʒ/ | Thể thực khuẩn |
| 32 | Electrophoresis | /ɪˌlɛktroʊfəˈriːsɪs/ | Điện di |
| 33 | Southern blotting | /ˈsʌðərn ˈblɒtɪŋ/ | Thấm Southern |
| 34 | Agarose | /ˈæɡəˌroʊs/ | Agarose |
| 35 | Nitrocellulose | /ˌnaɪtroʊˈsɛljʊˌloʊs/ | Nitrocellulose |
| 36 | Nylon membranes | /ˈnaɪlɒn ˈmɛmbreɪnz/ | Màng nylon |
| 37 | Denatured DNA | /diːˈneɪtʃərd diː ɛn eɪ/ | DNA biến tính |
| 38 | DNA probe | /diː ɛn eɪ proʊb/ | Mẫu dò DNA |
| 39 | Nucleotide sequence | /ˈnjuːkliəˌtaɪd ˈsiːkwəns/ | Trình tự nucleotide |
| 40 | Radioactive phosphorus | /ˌreɪdioʊˈæktɪv ˈfɒsfərəs/ | Phốt pho phóng xạ |
| 41 | Chemiluminescent moiety | /ˌkɛmɪˌluːmɪˈnɛsənt ˈmɔɪəti/ | Gốc phát quang hóa học |
| 42 | Hybridization | /ˌhaɪbrɪdaɪˈzeɪʃən/ | Lai hóa |
| 43 | Nucleic acid bases | /njuːˌkleɪɪk ˈæsɪd ˈbeɪsɪz/ | Base acid nucleic |
| 44 | Adenine (A) | /ˈædɪˌniːn/ | Adenine (A) |
| 45 | Thymidine (T) | /ˈθaɪmɪˌdiːn/ | Thymidine (T) |
| 46 | Guanine (G) | /ˈɡwɑːniːn/ | Guanine (G) |
| 47 | Cytosine (C) | /ˈsaɪtəˌsiːn/ | Cytosine (C) |
| 48 | X-ray film | /ˈɛks reɪ fɪlm/ | Phim X-quang |
| 49 | Radioautography | /ˌreɪdioʊɔːˈtɒɡrəfi/ | Chụp tự ghi phóng xạ |
| 50 | Autoradiography | /ˌɔːtoʊˌreɪdiˈɒɡrəfi/ | Chụp tự ghi phóng xạ |
| 51 | Single copy gene | /ˈsɪŋɡəl ˈkɒpi dʒiːn/ | Gen đơn bản |
| 52 | Restriction fragment length polymorphism (RFLP) | /rɪˈstrɪkʃən ˈfræɡmənt lɛŋkθ ˌpɒlɪˈmɔːrfɪzəm/ | Đa hình chiều dài đoạn cắt giới hạn |
| 53 | Alleles | /əˈliːlz/ | Alen (Allele) |
| 54 | Paternal alleles | /pəˈtɜːrnl əˈliːlz/ | Alen từ cha |
| 55 | Maternal alleles | /məˈtɜːrnl əˈliːlz/ | Alen từ mẹ |
| 56 | Autosomal recessive disease | /ˌɔːtəˈsoʊməl rɪˈsɛsɪv dɪˈziːz/ | Bệnh lặn trên nhiễm sắc thể thường |
| 57 | Homozygous | /ˌhoʊmoʊˈzaɪɡəs/ | Đồng hợp tử |
| 58 | Heterozygous | /ˌhɛtəroʊˈzaɪɡəs/ | Dị hợp tử |
| 59 | Carrier | /ˈkæriər/ | Người mang gen |
| 60 | Kilobases (kb) | /ˈkɪloʊˌbeɪsɪz/ | Kilobase (kb) |
| 61 | Human leukocyte antigen (HLA) | /ˈhjuːmən ˈluːkəˌsaɪt ˈæntɪdʒən/ | Kháng nguyên bạch cầu người (HLA) |
| 62 | Genomic DNA | /dʒəˈnoʊmɪk diː ɛn eɪ/ | DNA bộ gen |
| 63 | Pedigree | /ˈpɛdɪˌɡriː/ | Phả hệ |
| 64 | Oligonucleotides | /ˌɒlɪɡoʊˈnjuːkliəˌtaɪdz/ | Oligonucleotide |
| 65 | Primers | /ˈpraɪmərz/ | Mồi |
| 66 | DNA template | /diː ɛn eɪ ˈtɛmplɪt/ | Khuôn DNA |
| 67 | Taq polymerase | /tæk pəˈlɪməreɪz/ | Taq polymerase |
| 68 | Thermophilus aquaticus | /θərˈmɒfɪləs əˈkwætɪkəs/ | Thermophilus aquaticus |
| 69 | Thermocycler | /ˈθɜːrmoʊˌsaɪklər/ | Máy luân nhiệt |
| 70 | Cross-contamination | /krɒs kənˌtæmɪˈneɪʃən/ | Nhiễm chéo |
| 71 | False positive result | /fɔːls ˈpɒzətɪv rɪˈzʌlt/ | Kết quả dương tính giả |
| 72 | Aerosolization | /ˌɛəroʊˌsɒlaɪˈzeɪʃən/ | Sự tạo aerosol |
| 73 | SRY gene | /ɛs ɑːr waɪ dʒiːn/ | Gen SRY |
| 74 | Y chromosome | /waɪ ˈkroʊməˌsoʊm/ | Nhiễm sắc thể Y |
| 75 | Turner syndrome | /ˈtɜːrnər ˈsɪndroʊm/ | Hội chứng Turner |
| 76 | Karyotype | /ˈkærioʊˌtaɪp/ | Kiểu nhân đồ |
| 77 | Chromosomal gender | /ˌkroʊməˈsoʊməl ˈdʒɛndər/ | Giới tính nhiễm sắc thể |
| 78 | Sexual ambiguity | /ˈsɛkʃuəl ˌæmbɪˈɡjuːəti/ | Giới tính không rõ ràng |
| 79 | Complementary DNA (cDNA) | /ˌkɒmplɪˈmɛntəri diː ɛn eɪ/ | DNA bổ sung (cDNA) |
| 80 | Noncoding sequences | /ˌnɒnˈkoʊdɪŋ ˈsiːkwənsɪz/ | Trình tự không mã hóa |
| 81 | Regulatory elements | /ˈrɛɡjʊləˌtɔːri ˈɛlɪmənts/ | Yếu tố điều hòa |
| 82 | Alternate splicing | /ˈɔːltərnət ˈsplaɪsɪŋ/ | Cắt nối thay thế |
| 83 | Methylation | /ˌmɛθɪˈleɪʃən/ | Methyl hóa |
| 84 | Epigenetic changes | /ˌɛpɪdʒəˈnɛtɪk ˈtʃeɪndʒɪz/ | Thay đổi ngoại di truyền |
| 85 | Coding sequences | /ˈkoʊdɪŋ ˈsiːkwənsɪz/ | Trình tự mã hóa |
| 86 | Transcription | /trænˈskrɪpʃən/ | Phiên mã |
| 87 | Northern blotting | /ˈnɔːrðərn ˈblɒtɪŋ/ | Thấm Northern |
| 88 | Formaldehyde | /fɔːrˈmældɪˌhaɪd/ | Formaldehyde |
| 89 | Bacterial plasmids | /bækˈtɪəriəl ˈplæzmɪdz/ | Plasmid vi khuẩn |
| 90 | Solution hybridization | /səˈluːʃən ˌhaɪbrɪdaɪˈzeɪʃən/ | Lai hóa trong dung dịch |
| 91 | Quantitative reverse transcriptase-PCR (qRT-PCR) | /ˈkwɒntɪˌteɪtɪv rɪˈvɜːrs trænˈskrɪpteɪz piː siː ɑːr/ | PCR phiên mã ngược định lượng |
| 92 | Reverse transcriptase (RT) | /rɪˈvɜːrs trænˈskrɪpteɪz/ | Enzyme phiên mã ngược (RT) |
| 93 | Moloney murine leukemia virus (MMLV) | /məˈloʊni ˈmjʊəriːn luːˈkiːmiə ˈvaɪrəs/ | Virus bệnh bạch cầu chuột Moloney |
| 94 | Avian myeloblastosis virus (AMV) | /ˈeɪviən ˌmaɪəloʊˈblæstoʊsɪs ˈvaɪrəs/ | Virus u tủy bào gia cầm |
| 95 | RNA-dependent DNA polymerase | /ɑːr ɛn eɪ dɪˈpɛndənt diː ɛn eɪ pəˈlɪməreɪz/ | DNA polymerase phụ thuộc RNA |
| 96 | Competitive RT-PCR | /kəmˈpɛtətɪv ɑːr tiː piː siː ɑːr/ | RT-PCR cạnh tranh |
| 97 | RNA expression vector | /ɑːr ɛn eɪ ɪkˈsprɛʃən ˈvɛktər/ | Vector biểu hiện RNA |
| 98 | TaqMan assay | /ˈtækˌmæn əˈseɪ/ | Xét nghiệm TaqMan |
| 99 | Fluorescent 5′ nuclease | /flʊəˈrɛsənt faɪv praɪm ˈnjuːklieɪz/ | 5′ nuclease huỳnh quang |
| 100 | Reporter dye | /rɪˈpɔːrtər daɪ/ | Thuốc nhuộm báo cáo |
| 101 | Quencher dye | /ˈkwɛntʃər daɪ/ | Thuốc nhuộm dập tắt |
| 102 | Forster-type energy transfer | /ˈfɔːrstər taɪp ˈɛnərdʒi ˈtrænsfər/ | Truyền năng lượng kiểu Forster |
| 103 | Exonuclease | /ˌɛksoʊˈnjuːklieɪz/ | Exonuclease |
| 104 | Light Cycler | /laɪt ˈsaɪklər/ | Máy Light Cycler |
| 105 | Fluorogenic hydrolysis | /ˌflʊəroʊˈdʒɛnɪk haɪˈdrɒlɪsɪs/ | Thủy phân phát huỳnh quang |
| 106 | Fluorogenic hybridization probes | /ˌflʊəroʊˈdʒɛnɪk ˌhaɪbrɪdaɪˈzeɪʃən proʊbz/ | Mẫu dò lai hóa phát huỳnh quang |
| 107 | MicroRNA (miRNA) | /ˈmaɪkroʊ ɑːr ɛn eɪ/ | microRNA (miRNA) |
| 108 | Small interfering RNA (siRNA) | /smɔːl ˌɪntərˈfɪərɪŋ ɑːr ɛn eɪ/ | RNA can thiệp nhỏ (siRNA) |
| 109 | Transposons | /trænsˈpoʊzɒnz/ | Transposon (Gen nhảy) |
| 110 | Translational repression | /trænsˈleɪʃənəl rɪˈprɛʃən/ | Ức chế dịch mã |
| 111 | Gene silencing | /dʒiːn ˈsaɪlənsɪŋ/ | Làm câm lặng gen |
| 112 | 3′ untranslated region | /θriː praɪm ʌnˈtrænsleɪtɪd ˈriːdʒən/ | Vùng không dịch mã 3′ |
| 113 | Apoptosis | /ˌæpəpˈtoʊsɪs/ | Chết tế bào theo chương trình |
| 114 | Organogenesis | /ɔːrˌɡænoʊˈdʒɛnɪsɪs/ | Quá trình hình thành cơ quan |
| 115 | DiGeorge syndrome | /diːˈdʒɔːrdʒ ˈsɪndroʊm/ | Hội chứng DiGeorge |
| 116 | X-linked mental retardation | /ɛks-lɪŋkt ˈmɛntəl ˌriːtɑːrˈdeɪʃən/ | Chậm phát triển tâm thần liên kết X |
| 117 | Pathogenesis | /ˌpæθəˈdʒɛnɪsɪs/ | Cơ chế bệnh sinh |
| 118 | Deletions | /dɪˈliːʃənz/ | Mất đoạn |
| 119 | Insertions | /ɪnˈsɜːrʃənz/ | Chèn đoạn |
| 120 | Transpositions | /ˌtrænspəˈzɪʃənz/ | Chuyển vị |
| 121 | Single-base substitutions | /ˈsɪŋɡəl beɪs ˌsʌbstɪˈtjuːʃənz/ | Thay thế base đơn |
| 122 | High-throughput sequencing / Next-generation sequencing (NGS) | /haɪ-ˈθruːˌpʊt ˈsiːkwənsɪŋ / nɛkst-ˌdʒɛnəˈreɪʃən ˈsiːkwənsɪŋ/ | Giải trình tự thông lượng cao / Giải trình tự thế hệ mới (NGS) |
| 123 | Point mutation | /pɔɪnt mjuːˈteɪʃən/ | Đột biến điểm |
| 124 | Wild-type allele | /waɪld taɪp əˈliːl/ | Alen kiểu dại |
| 125 | Mutant allele | /ˈmjuːtənt əˈliːl/ | Alen đột biến |
| 126 | Pyrosequencing | /ˌpaɪroʊˈsiːkwənsɪŋ/ | Giải trình tự Pyro |
| 127 | Dideoxy method (Sanger) | /daɪˌdiːˈɒksi ˈmɛθəd/ | Phương pháp Dideoxy (Sanger) |
| 128 | Viral vectors | /ˈvaɪrəl ˈvɛktərz/ | Vector virus |
| 129 | Plasmid vectors | /ˈplæzmɪd ˈvɛktərz/ | Vector plasmid |
| 130 | Alkali treatment | /ˈælkəˌlaɪ ˈtriːtmənt/ | Xử lý bằng kiềm |
| 131 | 2′,3′-dideoxynucleoside triphosphates (ddNTPs) | /daɪˌdiːˌɒksiˈnjuːkliəˌsaɪd traɪˈfɒsfeɪts/ | Dideoxynucleoside triphosphate (ddNTPs) |
| 132 | Polyacrylamide electrophoresis | /ˌpɒliəˈkrɪləˌmaɪd ɪˌlɛktroʊfəˈriːsɪs/ | Điện di trên gel polyacrylamide |
| 133 | Capillary electrophoresis | /ˈkæpɪˌlɛri ɪˌlɛktroʊfəˈriːsɪs/ | Điện di mao quản |
| 134 | Microarray | /ˈmaɪkroʊəˌreɪ/ | Vi mảng (Microarray) |
| 135 | Expressed sequence-tags (ESTs) | /ɪkˈsprɛst ˈsiːkwəns tæɡz/ | Thẻ trình tự biểu hiện (ESTs) |
| 136 | Open reading frames (ORFs) | /ˈoʊpən ˈriːdɪŋ freɪmz/ | Khung đọc mở (ORFs) |
| 137 | Bioluminescence | /ˌbaɪoʊˌluːmɪˈnɛsəns/ | Phát quang sinh học |
| 138 | Apyrase | /ˈæpɪˌreɪz/ | Apyrase |
| 139 | ATP sulfurylase | /eɪ tiː piː ˌsʌlfəˈraɪleɪz/ | ATP sulfurylase |
| 140 | Luciferase | /luːˈsɪfəˌreɪz/ | Luciferase |
| 141 | Pyrophosphate | /ˌpaɪroʊˈfɒsfeɪt/ | Pyrophosphate |
| 142 | Adenosine 5′-phosphosulfate | /əˈdɛnəˌsiːn faɪv praɪm ˌfɒsfoʊˈsʌlfeɪt/ | Adenosine 5′-phosphosulfate |
| 143 | Luciferin | /luːˈsɪfərɪn/ | Luciferin |
| 144 | Oxyluciferin | /ˌɒksiˈluːsɪfərɪn/ | Oxyluciferin |
| 145 | Massively parallel sequencing | /ˈmæsɪvli ˈpærəˌlɛl ˈsiːkwənsɪŋ/ | Giải trình tự song song hàng loạt |
| 146 | Adapter ligation | /əˈdæptər laɪˈɡeɪʃən/ | Gắn adaptor |
| 147 | Flow cell | /floʊ sɛl/ | Flow cell (Buồng dòng chảy) |
| 148 | Bridge amplification | /brɪdʒ ˌæmplɪfɪˈkeɪʃən/ | Khuếch đại cầu |
| 149 | Reversible terminator-based method | /rɪˈvɜːrsəbl ˈtɜːrmɪˌneɪtər beɪst ˈmɛθəd/ | Phương pháp dựa trên chất kết thúc thuận nghịch |
| 150 | Personalized medicine | /ˈpɜːrsənəˌlaɪzd ˈmɛdɪsɪn/ | Y học cá nhân hóa |
| 151 | Immunohistochemistry (IHC) | /ɪˌmjuːnoʊˌhɪstoʊˈkɛmɪstri/ | Hóa mô miễn dịch (IHC) |
| 152 | Western blot | /ˈwɛstərn blɒt/ | Thấm Western |
| 153 | Protein lysate | /ˈproʊtiːn ˈlaɪseɪt/ | Dịch ly giải protein |
| 154 | Denaturing-gradient gel electrophoresis | /diːˈneɪtʃərɪŋ ˈɡreɪdiənt dʒɛl ɪˌlɛktroʊfəˈriːsɪs/ | Điện di trên gel gradient biến tính |
| 155 | Single-strand conformation polymorphism (SSCP) | /ˈsɪŋɡəl strænd ˌkɒnfɔːrˈmeɪʃən ˌpɒlɪˈmɔːrfɪzəm/ | Đa hình hình dạng sợi đơn (SSCP) |
| 156 | Dideoxy fingerprinting | /daɪˌdiːˈɒksi ˈfɪŋɡərˌprɪntɪŋ/ | Lấy dấu vân tay dideoxy |
| 157 | Heteroduplex mobility assay | /ˌhɛtəroʊˈdjuːplɛks moʊˈbɪləti əˈseɪ/ | Xét nghiệm di động dị hợp tử |
| 158 | High-performance liquid chromatography (HPLC) | /haɪ pərˈfɔːrməns ˈlɪkwɪd ˌkroʊməˈtɒɡrəfi/ | Sắc ký lỏng hiệu năng cao (HPLC) |
| 159 | CRISPR-Cas9 system | /ˈkrɪspər kæs naɪn ˈsɪstəm/ | Hệ thống CRISPR-Cas9 |
| 160 | Guide RNA (gRNA) | /ɡaɪd ɑːr ɛn eɪ/ | RNA dẫn đường (gRNA) |
| 161 | Somatic cells | /soʊˈmætɪk sɛlz/ | Tế bào soma (thân) |
| 162 | Germline cells | /ˈdʒɜːrmlaɪn sɛlz/ | Tế bào dòng mầm |
| 163 | Off-target alterations | /ɒf ˈtɑːrɡɪt ˌɔːltəˈreɪʃənz/ | Thay đổi ngoài mục tiêu |
| 164 | Positional genetics | /pəˈzɪʃənəl dʒəˈnɛtɪks/ | Di truyền học vị trí |
| 165 | Candidate gene approach | /ˈkændɪdət dʒiːn əˈproʊtʃ/ | Phương pháp tiếp cận gen ứng cử viên |
| 166 | Congenital adrenal hyperplasia (CAH) | /kənˈdʒɛnɪtəl əˈdriːnəl ˌhaɪpərˈpleɪʒə/ | Tăng sản tuyến thượng thận bẩm sinh |
| 167 | Reverse genetics | /rɪˈvɜːrs dʒəˈnɛtɪks/ | Di truyền học ngược |
| 168 | Positional cloning | /pəˈzɪʃənəl ˈkloʊnɪŋ/ | Dòng hóa vị trí |
| 169 | Human Genome Project (HGP) | /ˈhjuːmən ˈdʒiːnoʊm ˈprɒdʒɛkt/ | Dự án Bộ gen Người (HGP) |
| 170 | Segregation analysis | /ˌsɛɡrɪˈɡeɪʃən əˈnælɪsɪs/ | Phân tích phân ly |
| 171 | X-linked inheritance | /ɛks-lɪŋkt ɪnˈhɛrɪtəns/ | Di truyền liên kết X |
| 172 | Complex inheritance | /ˈkɒmplɛks ɪnˈhɛrɪtəns/ | Di truyền phức tạp |
| 173 | Phenotyping | /ˈfiːnoʊˌtaɪpɪŋ/ | Xác định kiểu hình |
| 174 | Autosomal inheritance | /ˌɔːtəˈsoʊməl ɪnˈhɛrɪtəns/ | Di truyền trên nhiễm sắc thể thường |
| 175 | Recessive trait | /rɪˈsɛsɪv treɪt/ | Tính trạng lặn |
| 176 | Dominant trait | /ˈdɒmɪnənt treɪt/ | Tính trạng trội |
| 177 | Penetrance | /ˈpɛnɪtrəns/ | Độ xâm nhập |
| 178 | Linkage analysis | /ˈlɪŋkɪdʒ əˈnælɪsɪs/ | Phân tích liên kết |
| 179 | Polymorphic markers | /ˌpɒlɪˈmɔːrfɪk ˈmɑːrkərz/ | Dấu hiệu đa hình |
| 180 | Meiosis | /maɪˈoʊsɪs/ | Giảm phân |
| 181 | Recombination frequency | /ˌriːˌkɒmbɪˈneɪʃən ˈfriːkwənsi/ | Tần số tái tổ hợp |
| 182 | Logarithm of odds (LOD) score | /ˈlɒɡərɪðəm əv ɒdz skɔːr/ | Điểm số LOD (logarit của tỷ lệ odds) |
| 183 | GenBank | /ˈdʒɛnˌbæŋk/ | GenBank |
| 184 | ENSEMBL | /ɑːnˈsɑːmbəl/ | ENSEMBL |
| 185 | Online Mendelian Inheritance in Man (OMIM) | /ˈɒnˌlaɪn mɛnˈdiːliən ɪnˈhɛrɪtəns ɪn mæn/ | Di truyền Mendel ở Người Trực tuyến |
| 186 | Sequence-tagged sites (STSs) | /ˈsiːkwəns tæɡd saɪts/ | Vị trí được đánh dấu bằng trình tự |
| 187 | Genomic clone | /dʒəˈnoʊmɪk kloʊn/ | Dòng vô tính bộ gen |
| 188 | Physical mapping | /ˈfɪzɪkəl ˈmæpɪŋ/ | Lập bản đồ vật lý |
| 189 | In vitro translation | /ɪn ˈviːtroʊ trænsˈleɪʃən/ | Dịch mã trong ống nghiệm |
| 190 | Loss-of-heterozygosity (LOH) | /lɒs əv ˌhɛtəroʊˌzaɪˈɡɒsəti/ | Mất dị hợp tử (LOH) |
| 191 | Comparative genomic hybridization | /kəmˈpærətɪv dʒəˈnoʊmɪk ˌhaɪbrɪdaɪˈzeɪʃən/ | Lai so sánh bộ gen |
| 192 | Von Hippel-Lindau disease (VHL) | /vɒn ˈhɪpəl ˈlɪndaʊ dɪˈziːz/ | Bệnh Von Hippel-Lindau |
| 193 | MEN1 (menin) | /ɛm iː ɛn wʌn (ˈmiːnɪn)/ | MEN1 (menin) |
| 194 | Cowden disease (PTEN) | /ˈkaʊdən dɪˈziːz (piː tɛn)/ | Bệnh Cowden (PTEN) |
| 195 | Peutz-Jeghers syndrome (STK11/LKB1) | /pɔɪts ˈjɛɡərz ˈsɪndroʊm/ | Hội chứng Peutz-Jeghers |
| 196 | Carney complex (PRKAR1A) | /ˈkɑːrni ˈkɒmplɛks/ | Phức hợp Carney |
| 197 | Whole-genome sequencing (WGS) | /hoʊl ˈdʒiːnoʊm ˈsiːkwənsɪŋ/ | Giải trình tự toàn bộ bộ gen (WGS) |
| 198 | Whole exome sequencing (WES) | /hoʊl ˈɛksoʊm ˈsiːkwənsɪŋ/ | Giải trình tự toàn bộ exome (WES) |
| 199 | Proband | /ˈproʊˌbænd/ | Bệnh nhân khởi phát |
| 200 | Loss-of-function mutations | /lɒs əv ˈfʌŋkʃən mjuːˈteɪʃənz/ | Đột biến mất chức năng |
| 201 | White Addison disease (WAD) | /waɪt ˈædɪsənz dɪˈziːz/ | Bệnh Addison trắng (WAD) |
| 202 | Primary adrenal insufficiency (PAI) | /ˈpraɪməri əˈdriːnəl ˌɪnsəˈfɪʃənsi/ | Suy thượng thận nguyên phát |
| 203 | Pigmentation | /ˌpɪɡmɛnˈteɪʃən/ | Sắc tố da |
| 204 | Melanocortin 1 receptor (MC1R) | /məˈlænoʊˌkɔːrtɪn wʌn rɪˈsɛptər/ | Thụ thể Melanocortin 1 (MC1R) |
| 205 | ACTH receptor (MC2R) | /eɪ siː tiː eɪtʃ rɪˈsɛptər (ɛm siː tuː ɑːr)/ | Thụ thể ACTH (MC2R) |
| 206 | α-melanocyte-stimulating hormone (α-MSH) | /ˈælfə məˈlænoʊˌsaɪt ˈstɪmjʊˌleɪtɪŋ ˈhɔːrmoʊn/ | Hormone kích thích tế bào hắc tố α |
| 207 | Proopiomelanocortin (POMC) | /proʊˌoʊpioʊməˈlænoʊˌkɔːrtɪn/ | Proopiomelanocortin (POMC) |
| 208 | Melanogenesis | /ˌmɛlənəˈdʒɛnɪsɪs/ | Quá trình tạo melanin |
| 209 | Eumelanin | /juːˈmɛlənɪn/ | Eumelanin |
| 210 | Hypopigmentation | /ˌhaɪpoʊˌpɪɡmɛnˈteɪʃən/ | Giảm sắc tố |
| 211 | Cutaneous cancers | /kjuːˈteɪniəs ˈkænsərz/ | Ung thư da |
| 212 | Melanoma | /ˌmɛləˈnoʊmə/ | U hắc tố |
| 213 | Familial glucocorticoid deficiency (FGD) | /fəˈmɪliəl ˌɡluːkoʊˈkɔːrtɪˌkɔɪd dɪˈfɪʃənsi/ | Suy giảm glucocorticoid gia đình |
| 214 | ACTH resistance | /eɪ siː tiː eɪtʃ rɪˈzɪstəns/ | Kháng ACTH |
| 215 | Autosomal recessive (AR) | /ˌɔːtəˈsoʊməl rɪˈsɛsɪv/ | Lặn trên nhiễm sắc thể thường (AR) |
| 216 | Allgrove syndrome | /ˈælɡroʊv ˈsɪndroʊm/ | Hội chứng Allgrove |
| 217 | Hydrocortisone | /ˌhaɪdroʊˈkɔːrtɪˌsoʊn/ | Hydrocortisone |
| 218 | Systems biology | /ˈsɪstəmz baɪˈɒlədʒi/ | Sinh học hệ thống |
| 219 | Pathophysiology | /ˌpæθoʊˌfɪziˈɒlədʒi/ | Sinh lý bệnh |
| 220 | Karyotyping | /ˌkærioʊˈtaɪpɪŋ/ | Lập kiểu nhân đồ |
| 221 | Metaphase | /ˈmɛtəˌfeɪz/ | Kỳ giữa (nguyên phân) |
| 222 | Anaphase | /ˈænəˌfeɪz/ | Kỳ sau (nguyên phân) |
| 223 | Chromosome banding | /ˈkroʊməˌsoʊm ˈbændɪŋ/ | Nhuộm băng nhiễm sắc thể |
| 224 | Giemsa staining | /ˈɡiːmsə ˈsteɪnɪŋ/ | Nhuộm Giemsa |
| 225 | Quinacrine | /ˈkwɪnəˌkriːn/ | Quinacrine |
| 226 | DAPI | /ˈdæpi/ | DAPI |
| 227 | Prophase | /ˈproʊˌfeɪz/ | Kỳ đầu (nguyên phân) |
| 228 | Prometaphase | /ˌproʊˈmɛtəˌfeɪz/ | Kỳ trước giữa (nguyên phân) |
| 229 | Mitosis | /maɪˈtoʊsɪs/ | Nguyên phân |
| 230 | Colcemid | /ˈkɒlsəˌmɪd/ | Colcemid |
| 231 | Kinetochores | /kɪˈniːtəˌkɔːrz/ | Tâm động |
| 232 | Centrosomes | /ˈsɛntrəˌsoʊmz/ | Trung thể |
| 233 | Actinomycin D | /ˌæktɪnoʊˈmaɪsɪn diː/ | Actinomycin D |
| 234 | Ethidium bromide | /ɪˈθɪdiəm ˈbroʊmaɪd/ | Ethidium bromide |
| 235 | BrDU (Bromodeoxyuridine) | /ˌbroʊmoʊdiˌɒksiˈjʊərɪˌdiːn/ | BrDU |
| 236 | Prenatal diagnostics | /ˌpriːˈneɪtəl ˌdaɪəɡˈnɒstɪks/ | Chẩn đoán trước sinh |
| 237 | Translocations | /ˌtrænsləˈkeɪʃənz/ | Chuyển vị |
| 238 | Inversions | /ɪnˈvɜːrʒənz/ | Đảo đoạn |
| 239 | Spectral karyotyping (SKY) | /ˈspɛktrəl ˌkærioʊˈtaɪpɪŋ/ | Lập kiểu nhân đồ quang phổ (SKY) |
| 240 | Aneuploidy | /ˌænjuːˈplɔɪdi/ | Lệch bội |
| 241 | Fluorescent in situ hybridization (FISH) | /flʊəˈrɛsənt ɪn ˈsaɪtuː ˌhaɪbrɪdaɪˈzeɪʃən/ | Lai tại chỗ phát huỳnh quang (FISH) |
| 242 | Molecular cytogenetics | /məˈlɛkjʊlər ˌsaɪtoʊdʒəˈnɛtɪks/ | Di truyền học tế bào phân tử |
| 243 | Germline mutation | /ˈdʒɜːrmlaɪn mjuːˈteɪʃən/ | Đột biến dòng mầm |
| 244 | Somatic mutation | /soʊˈmætɪk mjuːˈteɪʃən/ | Đột biến soma (thân) |
| 245 | Genomic imprinting | /dʒəˈnoʊmɪk ɪmˈprɪntɪŋ/ | In dấu bộ gen |
| 246 | Prader-Willi syndrome | /ˈprɑːdər ˈwɪli ˈsɪndroʊm/ | Hội chứng Prader-Willi |
| 247 | Angelman syndrome | /ˈeɪndʒəlmən ˈsɪndroʊm/ | Hội chứng Angelman |
| 248 | Pseudohypoparathyroidism | /ˌsuːdoʊˌhaɪpoʊˌpærəˈθaɪrɔɪˌdɪzəm/ | Giả suy cận giáp |
| 249 | Albright hereditary osteodystrophy | /ˈɔːlbraɪt hɪˈrɛdɪˌtɛri ˌɒstioʊˈdɪstrəfi/ | Loạn dưỡng xương di truyền Albright |
| 250 | Russell-Silver syndrome | /ˈrʌsəl ˈsɪlvər ˈsɪndroʊm/ | Hội chứng Russell-Silver |
| 251 | Beckwith-Wiedemann syndrome | /ˈbɛkwɪθ ˈviːdəmən ˈsɪndroʊm/ | Hội chứng Beckwith-Wiedemann |
| 252 | Persistent hyperinsulinemic hypoglycemia | /pərˈsɪstənt ˌhaɪpərˌɪnsjʊlɪˈniːmɪk ˌhaɪpoʊɡlaɪˈsiːmiə/ | Hạ đường huyết tăng insulin máu kéo dài |
| 253 | Transient neonatal diabetes | /ˈtrænziənt ˌniːoʊˈneɪtəl ˌdaɪəˈbiːtiːz/ | Đái tháo đường sơ sinh thoáng qua |
| 254 | Expressivity | /ˌɛksprɛˈsɪvɪti/ | Độ biểu hiện |
| 255 | Phenocopy | /ˈfiːnoʊˌkɒpi/ | Kiểu hình giả |
| 256 | Familial hypocalciuric hypercalcemia | /fəˈmɪliəl ˌhaɪpoʊˌkælsɪˈjʊərɪk ˌhaɪpərˌkælˈsiːmiə/ | Tăng calci máu giảm calci niệu gia đình |
| 257 | Calcium-sensing receptor | /ˈkælsiəm ˈsɛnsɪŋ rɪˈsɛptər/ | Thụ thể cảm nhận calci |
| 258 | Hyperparathyroidism-jaw tumor syndrome | /ˌhaɪpərˌpærəˈθaɪrɔɪˌdɪzəm dʒɔː ˈtuːmər ˈsɪndroʊm/ | Hội chứng u hàm-cường cận giáp |
| 259 | MEN4 syndrome | /ɛm iː ɛn fɔːr ˈsɪndroʊm/ | Hội chứng MEN4 |
| 260 | Single-nucleotide polymorphisms (SNPs) | /ˈsɪŋɡəl ˈnjuːkliəˌtaɪd ˌpɒlɪˈmɔːrfɪzəmz/ | Đa hình nucleotide đơn (SNPs) |
| 261 | Electronic medical records (EMRs) | /ɪˌlɛkˈtrɒnɪk ˈmɛdɪkəl ˈrɛkɔːrdz/ | Hồ sơ y tế điện tử |
| 262 | Direct-to-consumer genetic testing (DCGT) | /dɪˈrɛkt tə kənˈsjuːmər dʒəˈnɛtɪk ˈtɛstɪŋ/ | Xét nghiệm di truyền trực tiếp đến người tiêu dùng |
| 263 | Dexamethasone (DEX) suppression test | /ˌdɛksəˈmɛθəˌsoʊn səˈprɛʃən tɛst/ | Nghiệm pháp ức chế Dexamethasone |
| 264 | Cytochrome P450 | /ˈsaɪtəˌkroʊm piː fɔːr ˈfɪfti/ | Cytochrome P450 |
| 265 | Noninvasive prenatal testing (NIPT) | /ˌnɒnɪnˈveɪsɪv ˌpriːˈneɪtəl ˈtɛstɪŋ/ | Xét nghiệm tiền sản không xâm lấn (NIPT) |
| 266 | Cell-free DNA (cfDNA) | /sɛl-friː diː ɛn eɪ/ | DNA tự do ngoại bào (cfDNA) |
| 267 | Trisomies | /ˈtraɪsoʊmiz/ | Tam nhiễm (Trisomy) |
| 268 | Amniocentesis | /ˌæmnioʊsɛnˈtiːsɪs/ | Chọc ối |
| 269 | Single nucleotide variants (SNVs) | /ˈsɪŋɡəl ˈnjuːkliəˌtaɪd ˈvɛəriənts/ | Biến thể nucleotide đơn (SNVs) |
| 270 | Indels (Insertions/Deletions) | /ˈɪndɛlz/ | Chèn/mất đoạn (Indels) |
| 271 | Stop codon | /stɒp ˈkoʊdɒn/ | Codon kết thúc |
| 272 | Variants of unknown significance (VUS) | /ˈvɛəriənts əv ʌnˈnoʊn sɪɡˈnɪfɪkəns/ | Biến thể chưa rõ ý nghĩa (VUS) |
| 273 | Structural variation (SV) | /ˈstrʌktʃərəl ˌvɛəriˈeɪʃən/ | Biến thể cấu trúc (SV) |
| 274 | Copy number variants (CNV) | /ˈkɒpi ˈnʌmbər ˈvɛəriənts/ | Biến thể số lượng bản sao (CNV) |
| 275 | Chromosomal rearrangements | /ˌkroʊməˈsoʊməl ˌriːəˈreɪndʒmənts/ | Sắp xếp lại nhiễm sắc thể |
| 276 | Mobile element insertions | /ˈmoʊbaɪl ˈɛlɪmənt ɪnˈsɜːrʃənz/ | Chèn đoạn di động |
| 277 | Tandem repeats | /ˈtændəm rɪˈpiːts/ | Đoạn lặp nối tiếp |
| 278 | Pathogenic variant | /ˌpæθəˈdʒɛnɪk ˈvɛəriənt/ | Biến thể gây bệnh |
| 279 | Benign variant | /bɪˈnaɪn ˈvɛəriənt/ | Biến thể lành tính |
| 280 | In silico data | /ɪn ˈsɪlɪkoʊ ˈdeɪtə/ | Dữ liệu in silico (mô phỏng) |
| 281 | Secondary findings | /ˈsɛkənˌdɛri ˈfaɪndɪŋz/ | Phát hiện thứ cấp |
| 282 | American College of Medical Genetics and Genomics (ACMGG) | /əˈmɛrɪkən ˈkɒlɪdʒ əv ˈmɛdɪkəl dʒəˈnɛtɪks ænd dʒəˈnoʊmɪks/ | Trường Cao đẳng Di truyền và Gen y học Hoa Kỳ |
| 283 | Liquid biopsy | /ˈlɪkwɪd ˈbaɪɒpsi/ | Sinh thiết lỏng |
| 284 | Fetal fraction | /ˈfiːtəl ˈfrækʃən/ | Phân suất thai nhi |
| 285 | Mosaicism | /moʊˈzeɪɪˌsɪzəm/ | Thể khảm |
| 286 | Postzygotic mutation | /ˌpoʊstzaɪˈɡɒtɪk mjuːˈteɪʃən/ | Đột biến sau hợp tử |
| 287 | GNAS gene | /dʒiː ɛn eɪ ɛs dʒiːn/ | Gen GNAS |
| 288 | Monogenic disorders | /ˌmɒnoʊˈdʒɛnɪk dɪsˈɔːrdərz/ | Rối loạn đơn gen |
| 289 | Mendelian disorders | /mɛnˈdiːliən dɪsˈɔːrdərz/ | Rối loạn di truyền Mendel |
| 290 | Pancreatectomy | /ˌpænkriəˈtɛktəmi/ | Phẫu thuật cắt bỏ tuyến tụy |
| 291 | Sulfonylurea | /ˌsʌlfəˌnɪljʊˈriːə/ | Sulfonylurea |
| 292 | Adrenoleukodystrophy | /əˌdriːnoʊˌluːkoʊˈdɪstrəfi/ | Loạn dưỡng chất trắng thượng thận |
| 293 | Autoimmune etiology | /ˌɔːtoʊɪˈmjuːn ˌiːtiˈɒlədʒi/ | Nguyên nhân tự miễn |
| 294 | Gene-editing | /dʒiːn ˈɛdɪtɪŋ/ | Chỉnh sửa gen |
| 295 | Base-editing | /beɪs ˈɛdɪtɪŋ/ | Chỉnh sửa base |
| 296 | Anti-sense oligonucleotide (ASO) | /ˈænti-sɛns ˌɒlɪɡoʊˈnjuːkliəˌtaɪd/ | Oligonucleotide đối nghĩa (ASO) |
| 297 | Autologous stem cell transplantation | /ɔːˈtɒləɡəs stɛm sɛl ˌtrænsplænˈteɪʃən/ | Ghép tế bào gốc tự thân |
| 298 | Spinal muscular atrophy | /ˈspaɪnəl ˈmʌskjʊlər ˈætrəfi/ | Teo cơ tủy sống |
| 299 | Recombinant DNA technology | /riːˈkɒmbɪnənt diː ɛn eɪ tɛkˈnɒlədʒi/ | Công nghệ DNA tái tổ hợp |
| 300 | Functional genomics | /ˈfʌŋkʃənəl dʒəˈnoʊmɪks/ | Di truyền học chức năng |
